これは生体細胞であり、分子コンピューターとしてプログラマーの観点からこの投稿で検討されます 。 情報を処理するための多くの最新の方法とアルゴリズムは、1980年代には登場しませんでしたが、数百万年前に登場したことを学びます。
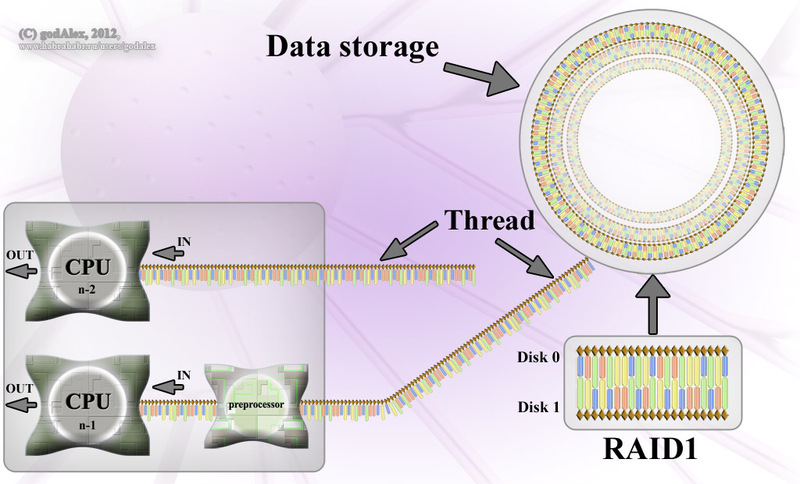
記憶媒体
データは分子記憶媒体-1つの分子(DNA、リング状の原核生物DNA)に保存され、クラスター(遺伝子)に分割されます。 データは4桁のコード(物理的には4つのヌクレオチドとして表されます:アデニン、チミン、シトシン、グアニン)。 RAID 1テクノロジーを使用して、高い耐障害性とデータ整合性が実現されます(DNAは二重らせんで、両方のらせんのヌクレオチドはペアA = T、C = Gで接続されています)。 データの整合性チェックは定期的に(DNAリガーゼタンパク質を使用して)実行され、見つかった損傷はもちろん保存された複製で修復されます。
このようなデータベースの複製は、情報キャリアを物理的に分離する方法により、特別なサービス(レプリコムと呼ばれるタンパク質複合体)によって実行され、すぐにキャリアの1つの完全バックアップを作成し、復元中の情報の整合性をチェックし、損傷を修復します。 リソースを節約するために、レプリカのバックアップコピーの作成が遅れます( DNA複製を参照)。 レプリケーションプロセスの最後に、データの両方のコピーには、RAID 1仕様に準拠したバックアップが含まれます。
媒体は情報の記録( 逆転写 )もサポートしていますが、実際には記録はほとんど使用されず、通常はウイルス( レトロウイルス )を破壊するのが困難です。
メディアパラメータ:メモリサイズ:スケーラブル、レプリケーション速度:> 1 GB / s (正確ではない) 、読み取り/書き込み速度:??? Mb / s / ??? Mb / s
RAM
これは分子コンピューターであるため、RAMの量によって、データストレージに関与しない自由にアクセス可能な分子の数が決まります。 メモリが割り当てられると 、「無料の」分子がRAMに取り込まれ、情報をエンコードする順序でスレッドに配置されます。
記憶が解放されると、糸状分子が分離し、自由分子の体積が補充されます。
記憶媒体からのデータの読み取り
RAM内の媒体からデータを読み取る( DNA転写 )開始時に、媒体上の目的の場所( 結合部位 )を検索します 。 次に、読み取りはファイルの最後の記号( おそらく AAAAシーケンス)まで続きます。 結果として生じる糸状分子(RNAまたはmRNA)は、処理のために送信されます。
読み取りプロセスは並行して実行できます。
イベントプログラムの実行
読み取りプロセスの開始は、イベント( 特別なタンパク質の影響下)に従って実行されます。 イベントまたはイベントのグループが到着すると、メディア(DNA)の目的のデータ領域からサービス情報の読み取りが開始されます。これには、実行のために読み取られる他の領域へのリンクが含まれる場合があります(転写の開始は、DNAシーケンスとDNAの有無によって複雑なプロセスですさまざまなタンパク質因子)。
CPU
コンピューターには、ウィスカ分子(RNA)に保存されたコードを実行する多くのプロセッサーがあります。したがって、このタイプのコンピューターは、文字通りと比both的にマルチスレッドをサポートしています。
誤って、かつて英語から翻訳されたコンピューターの「ストリーム」という言葉。 実際、翻訳リテラルスレッドは-スレッドになります!
マシンワードは3ビットですが、4進数です。 固定長のコマンドはすべて1マシンワード( コドン )です。 64種類のチーム。
注:プロセッサーに入る前に、ストリーム(RNA)の前処理( スプライシング )が行われ、余分なコマンドが削除されます。
プロセッサは、すべての物理的作業を行うタンパク質を作成することで分子コンピューターを制御します:新しい構造の構築、形状と動きの変更から新しいプログラムの起動(つまり、新しいRNAを読み取るコマンド)。
プロセッサアーキテクチャ: 64コマンド、実行速度:データバス(分子の配信速度)に依存します。
アセンブラー:どのチームですか?
チーム64、そのうち:
アセンブリ情報のエンコード:61(ただし、アクションは複製されます)
コントローラー:3(条件付き停止:UAG、UGA、強制停止UAA)、
予約済み:プロセッサがわずかに変更された場合、約41を他の目的に使用できます( 非正規コドンを参照)。
モジュラーコード構造
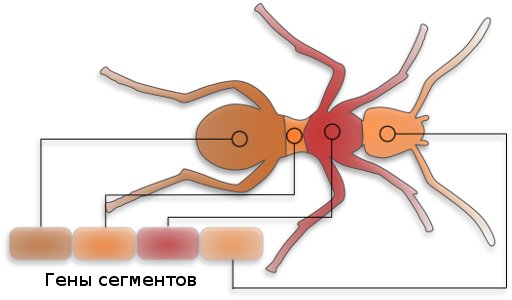
「アセンブラ」の形式のコードは上記で検討されましたが、今度は超高級言語の形式で検討します。 各遺伝子は特定の機能または機能のセットに関与していることが判明しました。
多細胞生物には、目、足などの生物の特定のシステムを構築する遺伝子があります。頭部、胴体、尾部などのセグメントを構築する遺伝子があります。
目の内部構造に関与する遺伝子の変化は、その内部構造にのみ影響し、目の位置に関与する遺伝子の変化は、目が体内に存在することにつながりますが、それらは他の場所から成長します。 目の位置の原因となる遺伝子を削除すると、それらは成長しません。
これらの「ブロック」(遺伝子)がほとんどの生物で交換可能であることは注目に値します。 そこで、科学者は実験を行いました。目の位置の原因となる遺伝子がショウジョウバエのハエから取り除かれました-新しいハエは目が見えませんでした。 ハエの次世代では、マウスから取った目の位置の原因となる遺伝子が挿入され、マウスではなく飛ぶ目が現れました。
多くの遺伝子はほとんどの生物で同じであるようであり、最も重要なことには、交換可能です!
遺伝子工学とは何ですか?
科学者は、いくつかの生物から遺伝子を分離し、それらを他の生物に挿入し、新しい生物を取得する方法をすでに学んでいます。 このプロセスは遺伝子工学と呼ばれます。 私の知る限り、遺伝子機能は試行錯誤によって検出されています:遺伝子に損傷を与え、変化したものを見て、「変異した」個人を見て、その遺伝子を参照のものと比較します(デバッガーでプログラムをクラックするようなもの)。
多くの生物でどの遺伝子が原因であるかを理解するとき、既製の遺伝子をコピーするだけでなく、まったく新しい遺伝子を作成することを学ぶとき、彼らは機械的なナノロボットを作成する必要はありません-それらはすでに生物学的にプログラムされた細胞の形になります。
結論として
最初の「DNAコンパイラ」は、クレイグベンター博士が率いる科学者グループによって2010年に作成されました。
また、ケンブリッジ大学の専門家によって保存され、DNAの抽象的な情報(サイズが約600キロバイトのファイルであり、これに制限はありません)が読み取られました。 しかし、他の記事でそれについて読むことができます。
ソースとリンク:
ウィキペディア:そこにあるすべての用語を調べます。 ただし、信頼と検証-明らかに不十分な数値が多すぎます。たとえば、この記事のコピー速度は、情報不足による非常に大きな誤差で推定されました。
ドキュメンタリーのビデオと映画:
「Creating Synthetic Life」2010-コンピューター上でDNAコードを編集し、生きた細胞(Craig Venter博士のグループ)が存在する間に生きた細胞を読み書きします。
「BBC:Cell」2009、「BBC:The Evolution of Life」-一部のシリーズでは、ショウジョウバエを用いた実験で、体の部位の位置に遺伝子がどのように関与するかが示されています。
「セルの内側の生活」ビデオ8分間、YouTubeで-「ナビゲーションプログラム」の実装をさらに説明せずに...
UPD :この記事は、2014年2月15日にCreative Commons CC-BY-NCでライセンスされています。