したがって、ここでは、この問題の解決を妨げる別の問題を簡単な言語で述べようとします。 そして、この問題は、私が解決策に選んだアプローチだけでなく、折り畳み作業にも共通していると思われます。
RNAInSpaceソフトウェアで、RNAヘリックスを手動で「ねじる」機能を実現し、そのような回転のジオメトリと制限が明らかになりました。 しかし、以前の記事によると、このソフトウェアはあまり興味がなかったので、このソフトウェアの別のデモバージョンは紹介しません。 そして、私が得るものについて話しましょう。
読者との会話を維持するために、いくつかの重要なポイントについて説明します。
chupvlは すでに尋ねました。 実際の問題でソリューションをテストしましたか、それとも理論を解体するだけですか? そこで彼にこのように説明しました:
私はリボザイムを折り畳んでいます。 リボザイムは、1つのクラスに区別されるため、同じ基本構造を持っています。 比較のために、実際に利用可能な別の2QUSリボザイムを使用します。 次に、違いと類似点を確認することが重要です。
コンクリート折りたたみの例
そして今、私は折りたたんでいるRNAがどのように見えるかを明確に示したいと思います。
以下はRNAの「骨格」で、これをベース(2QUS)として使用します。

三次構造が不明なウイロイドリボザイムNC_003540を崩壊させようとしています。 しかし、それは基本的なものにいくらか似ているはずです。 プライマリシーケンスはほぼ80%異なりますが。 以下は、私が手に入れた最高のオプションの1つです。 (自動ではなく、半自動-FoldItゲームを別の方法でのみプレイするかのように、完全自動化の前に-月のように)

ここでどのような違いを見ることができますか?
1.塩基配列(2QUS)はウイロイドリボザイムよりもわずかに長いため、ウイロイドリボザイムのRNAの末端は基本的な類似体のように長いらせんを形成しないことを理解する必要があります。
2.全体的なスタイリングは基本的に同じです。したがって、ウイロイドリボザイムは基本的に正しく折り畳まれています。
3.しかし、少なくとも1つの違いがあります。 ループL2(左下の図の領域を参照)-異なります。
3.a. このループには1つのヌクレオチドがあり、L1ループの他の2つのヌクレオチド(右下の図の領域)と4つの水素結合を形成します。 リボザイムループを一緒に保持するのはこれらの結合です。
(それがどのように見え、私がすでに解決した問題は何ですか-私は以前とここでコメントしまし た )
3.b. なぜ違いがあるのですか? ベースリボザイムでは、このループはウイロイドリボザイムIの研究よりも単純に長く、L2ループはL1ループとドッキングできるように構成を変更する必要があります。 そして、これはL2ループのかなり不自然な位置であるため、スパイラル自体の構成(左側の方が高い)はこれに合わせて変更する必要があります。
4.そして最後に、これらすべての問題を解決した後、私はリボザイムの端をリボザイム核の正しい位置に折り畳むことは問題にならないと考えて、手をこすりました。 しかし、彼は間違っていました。 らせんの端の間に必要な水素結合を形成することはできませんでした(図では、端が互いに十分に近くないことがわかります)。
なぜリボザイムを完成できなかったのですか?
ロールアップしたRNA構造の問題を比較し始めました。 両端が収束したくなかったのは、 それらは、スパイラルL1とL2を接続する突起によって防止されました(上から中央)。 そして、この突起が誤った位置になった後、文字通り1つまたは2つのヌクレオチドであることが判明しました。 彼らはモデリングにあまり注意を払っていませんでした。 それらは水素結合を形成しませんでした。 そして彼らは受け入れた-ランダム。 そして当然、ランダムな位置ではリボザイムが到達できませんでした。 (分岐:考えてみてください-現在使用されているほとんどの方法で、ヌクレオチドがランダムな位置に配置され、RNA分子全体の平均統計に従って正しいヌクレオチドが選択された場合、シミュレーション中にどうなりますか?)
すでに折りたたまれた構造でこの位置を変更しようとしました(2番目の図)。 しかし、これは不可能であることが判明しました-ほとんどすべての接続を再構築する必要がありました。 すでに形成されているすべての水素結合を破壊します。
そして、私は考えました。 私はウォットからのコメントを思い出しました: 初期状態は結果を決定し、以前
そして、初期位置自体が、どの「最終」状態が達成されるかを決定します。 この場合の初期状態は、チェーン自体を作成するプロセスです。 つまり、良い問題のためには、最初の問題を追加し、次の問題を追加し、システムを最小化し、次の問題を追加し、再度最小化する必要があります。
...
同様に、長いチェーン全体を考慮することは有害です-これは信じられない状態であり、信じられない状態とそうでない状態の両方を達成できますが、オプションのサイズが大きいため、2番目のものがはるかに多くなります。
これはかなり正しい発言であり、私にとっては、以前書いた私の科学論文の1つで知られていました。
...
ゲーム理論の用語では、これはゲームの初期位置から特定の最終状態に到達する可能性を保証する必要があることを意味します。 そして、例えば、チェスのゲームで、ゲームがどのように始まり、勝利条件が何であるかを知っていて、チェスのルールがゲームの始まりから終わりまでを確保することを知っているなら、ここで-高分子の折り畳みをモデル化するとき-そのようなルールを見つけて証明する必要があります彼らは折り畳みプロセスを提供すること。
したがって、そのようなアプローチは私の結果と矛盾し、ハーフロール状態からよりもチェーンに拡張された初期状態から最小化を開始する方がよいと一般的に答えました。
しかし、いわゆるスタッキング相互作用がまだあります。これは、単純に六角形と呼ばれるヌクレオチドがコインのスタックを形成するように配置されるときです。 下の図では、最初の6ヌクレオチド(下から数えて)がスタッキング相互作用にあります。
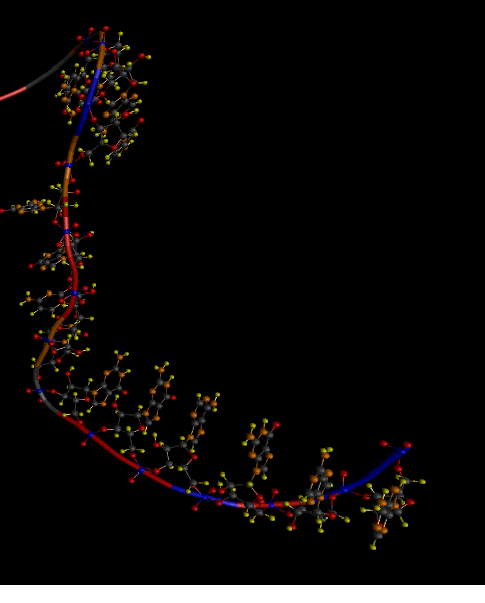
そのため、RNAが徐々に現れる場合、最初の水素結合が形成される前に、少なくとも20ヌクレオチドが現れる必要があります。
以前は、チェーンに引っ張られただけだと思っていました。 しかし、彼らは実際に次々と登場し、スタックの特徴的な位置を取ります。 チェーンは伸びているように見えますが、偶然ではありません。 そして、これは出発点にとって重要なことです。
しかし、もう1つ問題がありました。 DNAの二重鎖がどのように見えるかを思い出せば、その長さは非常に大きくなる可能性があります。 そして、まさにそのような積み重ねの相互作用があります。 しかし、RNAはこれを目指して努力していますが、ごく小さな領域にのみ十分であり、3番目の図では、7ヌクレオチドから始まるRNAチェーンの方向がどのように変わるかを示しています。
いくつかの結論
私がこれまでずっとリードしてきたことは、おそらくあまり明確ではありません。 それを理解してみましょう。
1.スタッキングは初期位置を作成するために重要であり、上記のシミュレーションでは、RNAが実際に折り畳まれているときに、自然に折りたたまれたときのような状況を防ぐのに役立ちます-ただし、いくつかのヌクレオチドはランダムな位置に残ります(スタッキングによって接続されません)-およびこれは、さらに折り畳むのを妨げます。
2. RNAスタッキングは、厳密ならせんが形成されるDNAほど安定ではありません。 RNAでは、いくつかの節点で、スタッキング力が連続するヌクレオチドのペア間で作用しなくなります。 そして、チェーンの方向が変わります。
3.鎖の方向が変化し、ヌクレオチドの相補的なペアが見つかると、水素結合が形成され始めます。これはスタッキングよりも強い安定化効果があります。
4.しかし、チェーンが成長し、2つ以上のらせんに分割されると、これらのらせんのループは非標準の水素結合によって相互接続されます。 これはさらに強力な相互作用であり、この時点で以前のスタッキングである水素結合の近似が崩壊する可能性があり、形状はらせんループから始まります。
これが仮説です。私が現時点で得た折りたたみモデルです。 まだ確定的に検証する必要がありますが、少なくとも公平ではない他の多くのバリエーション(階層モデル(最初に2次構造を作成し、次にそれらを3次構造に結合することを言う)など)が現在テストされています。
その結果、スタッキングのタスクは、必要な水素結合の形成を促進することであることがわかりました。 そして、その存在なしでは、水素結合自体は形成されません。
PS実際、すべてはもう少し複雑ですが、私はあなたをロードしたくありません。 そのため、彼は単純に約束したように見えますが、完全にはうまくいきませんでした。 しかし、私は何かが明確でない質問に答える準備ができています。