 私に知られているすべての技術科学および自然科学の中で、おそらく人々が最悪の考えを持っているのはバイオインフォマティクスについてです。 それは、ある程度まで間違っているか、まったくそうではありません。 2年前にバイオインフォマティクスを始めたとき、私自身はこの科学についてまったく知識がありませんでした。 時間が経つにつれて、私はバイオインフォマティクスが直面するタスク、それらが使用するもの、そして彼らの仕事から何が生じる可能性があるかをよりよく想像し始めました。
私に知られているすべての技術科学および自然科学の中で、おそらく人々が最悪の考えを持っているのはバイオインフォマティクスについてです。 それは、ある程度まで間違っているか、まったくそうではありません。 2年前にバイオインフォマティクスを始めたとき、私自身はこの科学についてまったく知識がありませんでした。 時間が経つにつれて、私はバイオインフォマティクスが直面するタスク、それらが使用するもの、そして彼らの仕事から何が生じる可能性があるかをよりよく想像し始めました。
バイオインフォマティクスには、試験管、試薬、細菌、白衣はありません。 彼らが持っている主なツールは、ラップトップ、紙付きのペン、またはマーカー付きのホワイトボードです-一般的に、すべてがプログラマーのようなものです。 作業自体はIT企業での作業と非常に似ており、研究室は小さな開発部門のようなものです。 そして、違いは何ですか? さて、私は答えようとします。
まず、タスクはほとんどアルゴリズムです。 つまり、プログラムを書く前に、いくつかの記事を読んで、自分で考え、同僚とアイデアを話し合ってから、実装を進める必要があります。 第二に、大量のデータを扱う必要があるため、実装は可能な限り効率的でなければなりません。 ただし、効果的で論理的で完全にデバッグされたプログラムでさえ、望ましい結果をもたらさない場合があります。 これの主な理由は、データの生物学的起源です。これは、膨大な数のエラーと、異なる研究所からのデータ間の大きな違いを意味します。
別の、おそらくバイオインフォマティクスとプログラミングの最も目に見える違いは、研究と出版です。 バイオインフォマティクスは科学です。つまり、世界で起こるすべてのことに注意する必要があります。 このために、多数の会議、他国の研究所とのコラボレーション、そしてもちろん出版物があります-あなたもあなたの成果について全員に伝える必要があります。 これらすべてがなければ、あなたは勤勉に車輪を再発明することができます。
一般に、バイオインフォマティクスの印象はそれだけですが、特に例があり、そう遠くないので、例を挙げて伝えるのが最善です。 しかし、まず最初に。
アルゴリズム生物学研究室
2010年、「メガグラント」プログラムがロシアで開始されました。 西洋の有力な科学者(ロシアを長く離れていたほとんどの場合)のリーダーシップの下で、新しい科学研究所が設立され始めました。 これらの1つは、彼の分野で最も有名な科学者の1人であるPavel Pevznerのリーダーシップの下、SPbAUのアルゴリズム生物学研究所でした。 パベルはモスクワ工科大学を卒業しましたが、その後すぐに米国に向かい、コンピューターサイエンス(正確にはバイオインフォマティクス)を取得し、現在はサンディエゴのカリフォルニア大学の教授を務めています。
研究室で彼らが何をしているのかを正確に伝える前に、読者に物事を紹介する価値があります。
ゲノミクスについて少し
すべての読者がゲノムという言葉を聞いたことがあると思います。 生物学者にとって、ゲノムはDNA分子、つまり細胞の核に折り畳まれた染色体に組織化された4つのヌクレオチドの長い鎖です。 ゲノムは4つの文字(A、C、G、T)で構成される文字列として表示されます。 ゲノムの長さは、数十億または数百億の文字に達する可能性があります。 生物学者は、遺伝子全体の読み方を知りません-最大150の「文字」の小さな断片でのみで、それでもエラーがあります。 私たちの仕事は、これらの断片から元のゲノムを復元すること、またはより頻繁に言うように、組み立てることです。
明確にするために、この比較をもたらすことができます:同一の新聞のパックを想像してください。 ここで、この束が破裂し、小さな紙片が飛び散ったり、混ざったり、劣化したり、完全に燃え尽きたりすることを想像してください。 そして、このゴミの山をさらに下って、元の新聞を接着したいと思います。
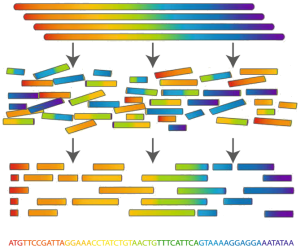 だから、それはゲノムにあります。 最初のテクノロジーにより、数千文字までのゲノムの断片を読み取ることができました。 これらの技術は信じられないほど高価でした-数十億ドルと世界中の数百人の研究室スタッフによる数年間のハードワークが、最初のヒトゲノムのアセンブリに費やされました。 最新のテクノロジーでは、短い断片を読み取ることができますが、1桁安く、大量に読み取ることができます。 もちろん、ギガバイトの入力データの処理は自動的に行われます。 この目的のために、ゲノムコレクターと呼ばれるプログラム、またはより頻繁に-アセンブラー(英語アセンブルから)が開発されています。 元のゲノムのいくつかの機能(繰り返し領域など)、および入力データの多数のエラーにより、コレクターの作業の結果はゲノム全体ではなく、ゲノムのかなり長いセクションにすぎません。 取得されたセクションが長いほど、元のゲノムとの類似性が高いほど、結果はより適切に考慮されます。
だから、それはゲノムにあります。 最初のテクノロジーにより、数千文字までのゲノムの断片を読み取ることができました。 これらの技術は信じられないほど高価でした-数十億ドルと世界中の数百人の研究室スタッフによる数年間のハードワークが、最初のヒトゲノムのアセンブリに費やされました。 最新のテクノロジーでは、短い断片を読み取ることができますが、1桁安く、大量に読み取ることができます。 もちろん、ギガバイトの入力データの処理は自動的に行われます。 この目的のために、ゲノムコレクターと呼ばれるプログラム、またはより頻繁に-アセンブラー(英語アセンブルから)が開発されています。 元のゲノムのいくつかの機能(繰り返し領域など)、および入力データの多数のエラーにより、コレクターの作業の結果はゲノム全体ではなく、ゲノムのかなり長いセクションにすぎません。 取得されたセクションが長いほど、元のゲノムとの類似性が高いほど、結果はより適切に考慮されます。
ゲノムアセンブリタスク
最も一般的なケースでゲノムを組み立てるタスクをとる場合、それは次のように定式化される最短スーパーストリング問題に過ぎません:与えられたセットの各ラインがそのサブストリングになるように最短ラインを見つけます。 このタスクはNP完全です。 しかし、同じ長さの元の文字列のすべての可能な部分文字列があると仮定すると、問題は多項式時間で解決できます。 ゲノムアセンブリはまさにそのような場合です。 2001年、Pavel Pevznerは、Count de Bruyneを使用したゲノムアセンブリへの効率的なアプローチを提案しました。 このアプローチの主なアイデアは、ほぼすべての現代のゲノムアセンブラーで使用されています。 ただし、実際には、前述の生物学的エラーによってすべてが非常に複雑になるため、主なタスクは、ゲノムのアセンブリ中に発生するさまざまなサブタスクのヒューリスティックを開発することです。
アルゴリズム生物学の研究室では、アセンブラーの開発に焦点を合わせることが決定されました。 もちろん、研究所の設立時には、膨大な数のゲノムコレクターがいました。 なぜ別のものを作成するのですか? 実際、アセンブリタスクは、一見したところよりもはるかに広範です。 生物学者は膨大な数の異なるタイプの入力データを作成しますが、それぞれの入力データは、その特異性を考慮した新しい方法の開発を必要とします。 さらに、ゲノムのアセンブリには多数のステップとアルゴリズムが含まれているため、現代のすべてのアセンブラは同じアプローチを使用していますが、結果は大きく異なる可能性があります。 研究室は、多くの点で既存のものよりも優れているアセンブラーを取得することを任されました。
バイオインフォマティクスへの道
私はバイオインフォマティクスに夢中になりました。 私はサンクトペテルブルク経済大学の行政で勉強しました。そして、すべての学生と同じように、学期の初めに研究を選択しなければなりませんでした。 新しい分野に挑戦するために、バイオインフォマティクスプロジェクトを選びました。 最初は、アルゴリズムを開発して実装するのではなく、生物学を勉強しなければならないかもしれないと心配しました。 しかし、幸いなことに、恐怖は実現しませんでした-この主題領域への没入は、他の場合と同じように起こります。 徐々に、あなたはより多くのことを理解し、新しいことを学び始めます。そして、たとえ生物学が学校で最も好きな科目であるとは程遠いとしても、それに対する関心は十分に早く現れます。 ほぼすぐに、バイオインフォマティクスがまさに私がやりたいことであることに気付きました-研究の要素と興味深い主題分野でのプログラミング。
私がプロジェクトを行っている間に、私が言及したアルゴリズム生物学の研究室が組織されました。 2011年の夏に、私はそのインターンシップを無事に完了し、常任研究員として残りました。 研究所全体について話すと、ゲノムの組み立て、西洋の研究所との協力、科学会議、新しいことを学ぶ絶え間ない機会、そしてもちろん非常に良いチームに限らず、膨大な数の興味深いプロジェクトがあります。
おそらく、研究室での仕事について、そして未だに多くの未解決の問題を抱えているバイオインフォマティクス全般について、そしてさまざまな問題における特定のアプローチとアルゴリズムについて、長い間話すことができるでしょう。 しかし、広大さを受け入れることは不可能であり、したがって、物語はあるものと次回についてのものになります。 そして何が正確に-あなたの願いに依存します。