
昨年、Arthur Kadurinと私は、ニューラルネットワークトレーニングの新しい波であるディープラーニングに参加することにしました。 機械学習は実際には多くの分野で使用されていないことがすぐに明らかになり、私たちは機械学習をどのように適用できるかを理解しています。 興味深い分野とその中の強力な専門家を見つけるために残った。 そこで、Insilico Medicine(Skolkovo FoundationのBMTクラスターの常駐)のチームとモスクワ物理学技術研究所の開発者に会い、癌に対する薬を見つける作業に協力することにしました。
以下の記事のレビューを読んでください:意味のあるリードの宝庫:腫瘍学における新しい分子開発のための深い敵対オートエンコーダーの適用 。これは、Insilico MedicineとMIPTの同僚とアメリカの雑誌Oncotargetで準備したもので、テンソルフローフレームワークでの提案モデルの実装に重点を置いています。 最初のタスクは次のとおりでした。 タイプのデータがあります:物質、濃度、癌細胞の成長指標。 特定の濃度で腫瘍の成長を止める新しい物質を生成する必要があります。 データセットはNCI Wiki Webサイトで入手できます。
データをさらに詳しく説明しましょう。
物質は、バイオインフォマティクスの標準形式で表示されます-SMILES、実際、これは化合物をASCIIで記録する統一された方法です。 残念ながら、完全な統一はありません;異なるパッケージは異なるSMILESを生成します。 CACTVSパッケージを使用して作成されたものを使用しました。 これに関する詳細は、例えばここに書かれています 。 各SMILESは、次元166:Molecular ACCess System(MACCS)化学フィンガープリントのバイナリ表現に変換できます。 この表現の各ビットは、対応する分子に関する質問への回答であり、MACCSキーは対応する質問のセットです。 質問の例:分子内の酸素原子は3個未満ですか? 分子にはサイズ3の環がありますか? より詳細な説明はこちらです。
成長指数は次のように定義されます。
どこで -初期腫瘍サイズ、 -薬物の存在下で特定の時間間隔後の腫瘍のサイズ、 -薬物を添加しない対照群の腫瘍のサイズ。 実際に 腫瘍の成長速度、または腫瘍の減少速度を示しています。
記載された治療の後、フィンガープリント、ログ(濃度)、GIの78 728トリプルが得られ、6252個の分子を記述します。 モデルを検証するために、Pubchemデータベース( The PubChem Project )のほぼ1億個の分子のバイナリ表現を使用しました。
受信したソースデータの例:
| 指紋 | ログ(濃度) | 成長阻害率 |
|---|---|---|
| 000011100010 ... | –5 | 10% |
| 00000110101 ... | –7 | –15% |
| 10010011000 ... | –4.8 | 75% |
解決策
素朴なアプローチ
頭に浮かぶ最初のアプローチ:リグレッサー(たとえば、xgboost)をトレーニングして、フィンガープリントから成長インジケーターを決定し、デュースを記録(濃度)し、大規模なデータベースからそのようなデュースをサンプリングし、最適なものを選択します。

このようなアプローチを良いものにもたらすことはできません。
ジェネレーティブアプローチ
別のオプション:ペア(指紋、濃度)の生成方法を学習し、化合物の大規模なベースから類似の分子を探す。 最初の質問の1つ:異なる分子を比較する方法? 幸いなことに(?)、このケースではジャカード測度を使用できることが実験的に判明しました。多くのアプリケーションでは、ジャカード係数が0.8より大きい分子はすでに近いと見なされています。
このような問題を解決することに加えて、新しい生成ニューラルネットワークアプローチを使用する学習の目標を追求しました。 VAE(variational autoencoder)とGAN(generalative adversarial encoder)の2つの最新の生成モデルが知られています。 両方のモデルがすでにHabréで何度か説明されているため、我々はそれらについての短い話に限定しています。
どこで -入力データ -潜在表現、および -モデルパラメーター。
標準VAEは配布を想定しています そして 通常の オートエンコーダー構造を持つニューラルネットワークを使用します。
入力データ エンコーダの入力に供給され、その出力は正規分布です 。 次に、この分布からベクトルがサンプリングされます 、デコーダの入力に供給されます。 出口で-ベクトル 再構成ベクトル 。
ニューラルネットワークをトレーニングする場合、2つの損失関数間の平均が最適化されます。潜在層の分布と および再構成エラー-間の距離 そして 。
GANの場合 、ジェネレーターとディスクリミネーターという 2つのニューラルネットワークがトレーニングされます。 ジェネレーターは潜在変数を変換します 事前配布からサンプリング 、入力データ空間に格納され、弁別器はサンプリングされたデータと実際のデータを区別することを学習します。 問題はゲーム理論の形式で定式化できます。

最後に、 Adversarial Autoencodersの記事では、VAEモデルとGANモデルの組み合わせを紹介しています。 通常のAEと同じ、入力 エンコーダに供給され、その出力 、順番に、デコーダーの入力に到達します。 ネットワーク弁別器はベクトルを区別することを学習します からサンプリング 、およびベクトル 。 GANと同様に、2つの損失関数が最適化されています。
- 発電機の重みは、識別器を「だます」ように最適化されています。 から 。
- 識別器の重みは区別するために最適化されています から 。
この場合、追加の損失関数-再構成エラーが導入されます。 損失関数による最適化が順番に行われます。 たとえば、最初のトレーニングサイクルは次のようになります。ミニバッチで弁別器をトレーニングし、次のミニバッチでエンコーダーをトレーニングし、3番目のミニバッチでオートエンコーダーをトレーニングします。

VAEモデルとGANモデルは最近、画像生成タスクで特に人気があり、GANベースのモデルは実際のパフォーマンスが向上しています(例: NIPS 2016 Tutorial:Generative Adversarial Networks )。
このため、および自動エンコーダーの潜在層に成長インデックスを追加するのが簡単であるため、特定の問題を解決するための後者のアプローチに決めました(異なるモデルの詳細な比較を含む新しい記事の公開を待っています)。 さらに、問題を解決する際に、安定して動作する標準のGANアーキテクチャのハイパーパラメーターを見つけることができませんでした。
到達したモデルは次のとおりです。
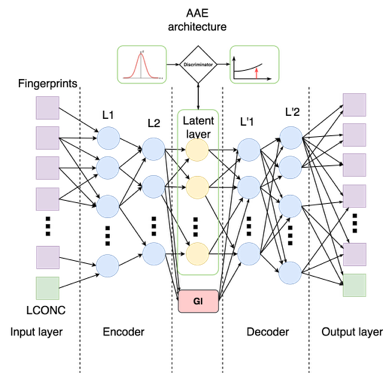
集中がエンコーダ出力にできるだけ影響を与えないようにするために、別の損失関数を追加しました-マニホールド損失:
どこで -エンコーダー、および -コサイン測定。
最後に、潜在層で回帰問題を解決します。指紋と濃度の潜在表現に従って、決定します 対応する損失関数を追加します。 その結果、各トレーニングステップは5つのステージで構成されます。
- トレーニング弁別器の重み(GANの場合と同様)。
- トレーニングジェネレーターの重み(GANなど)。
- 自動エンコーダトレーニング、すなわち再構成エラーの最適化。
- リグレッサートレーニング。
- 損失関数の最適化-多様体損失。
学習プロセス全体のコードがここにあります (現時点では、コードの実験を続けており、近い将来に改善されたバージョンを投稿する予定です)。
ネットワークアーキテクチャとメトリックの実装をさらに詳しく調べてみましょう。 個別に最適化できるように、ネットワークの各部分の重みを個別のtf.name_scopeで選択します。 さらに、入力層から濃度を別の変数に分離し、その再構成誤差を個別に考慮します。
通常、ネットワークは線形層とアクティベーション関数のシーケンスです。 たとえば、エンコーダーは次のように構成されます( aae_v3.py 、行109-127):
# Encoder net: 166+1->128->64->3+1 with tf.name_scope("encoder"): encoder = [self.visible_tensor] sizes = [self.input_space + 1, 128, 64, self.latent_space] for layer_number in xrange(encoder_layers): with tf.name_scope("encoder-%s" % layer_number): enc_l = layer_output(sizes[layer_number], sizes[layer_number + 1], encoder[-1], 'enc_l') encoder.append(enc_l) with tf.name_scope("encoder-fp"): self.encoded_fp = layer_output(sizes[-2], sizes[-1], encoder[-1], 'encoded_fp', batch_normed=False, activation_function=identity_function) with tf.name_scope("tgi-encoder"): self.encoded_tgi = layer_output(sizes[-2], 1, encoder[-1], 'encoded_tgi', batch_normed=False, activation_function=identity_function) self.encoded = tf.concat(1, [self.encoded_fp, self.encoded_tgi])
弁別器損失関数:
オーバーフローに対処するために、シグモイドを弁別器の出力ですぐにではなく、損失関数の内部で直接考慮します。 コード内:
self.disc_loss = tf.reduce_mean(tf.nn.relu(self.disc_prior) - self.disc_prior + tf.log(1.0 +tf.exp(-tf.abs(self.disc_prior)))) + tf.reduce_mean(tf.nn.relu(self.disc_enc) + tf.log(1.0 + tf.exp(-tf.abs(self.disc_enc))))
エンコーダー損失関数:
コード内:
self.enc_fp_loss = tf.reduce_mean(tf.nn.relu(self.disc_enc) - self.disc_enc + tf.log(1.0 + tf.exp(-tf.abs(self.disc_enc))))
自動エンコーダ損失機能:
コード内:
self.dec_fp_loss = tf.reduce_mean(tf.nn.sigmoid_cross_entropy_with_logits(self.decoded_fp, self.fingerprint_tensor)) self.dec_conc_loss = tf.reduce_mean(tf.square(tf.sub(self.conc_tensor, self.decoded_conc))) self.dec_loss = self.dec_fp_loss + self.dec_conc_loss
リグレッサー:
コード内:
self.enc_tgi_loss = tf.reduce_mean(tf.square(tf.sub(self.tgi_tensor, self.encoded_tgi)))
コードのマニホールド損失:
fp_norms = tf.sqrt(tf.reduce_sum(tf.square(self.encoded_fp), keep_dims=True, reduction_indices=[1])) normalized_fp = tf.div(self.encoded_fp, fp_norms) cosines_fp = tf.matmul(normalized_fp, tf.transpose(normalized_fp)) self.manifold_cost = tf.reduce_mean(1 - tf.boolean_mask(cosines_fp, self.targets_tensor))
私たちの実験では、異なる構成のネットワークは不安定に動作しました。 メイン学習プロセスの前に安定性を高めるために、ディスクリミネーターとエンコーダーが平衡に達するまでステップ1、2、および5を繰り返しました:discriminator_loss <0.7およびencoder_loss <0.7。
モデル検証
トレーニング後、GANの条件で標準的な方法で1000ペア(指紋、濃度)のランダムサンプルを生成します :
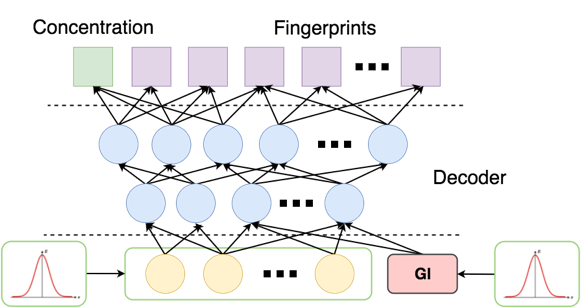
潜在層に事前分布を与え、代わりに -通常のノイズのある小さな数字 (ゼロは、関数が この時点でギャップがあります)。 出力では、濃度<-5の指紋ビットの32の確率ベクトルが得られました。
最後に、得られたベクターと同様に、大きな塩基から実際の分子を見つけることが残っています。 少なくとも3つのアプローチがあります。
- NLL(ログの信頼性)を数える
- 得られた分布から多次元ベルヌーイ分布としてサンプリングし、ジャカード距離を計算します。
- 0.5のカットオフを設定して、ジャカードの距離を計算します。
各確率ベクトルについて、3つのアプローチのそれぞれについて上位10個の「類似」分子を見つけ、同様の結果を得ました。
結果
その結果、トレーニングセットにない69のユニークな物質が見つかりました。 The PubChem Projectデータベースによると、約半分の物質がすでに癌に対して使用されているか、癌性腫瘍に対して効果が証明されています。 残りの半分の物質も潜在的に癌と戦うことができると期待しています。
したがって、比較的小さな入力データを使用した比較的単純なアプローチにより、良い結果を得ることができました。 同様の方法で多くのバイオインフォマティクスの問題を解決できると期待されています。
残念ながら、それぞれの場合で、競合するネットワークは異なる動作をし、ハイパーパラメーターの選択を間違えると、誤った決定に収束します(ジェネレーターがディスクリミネーターを無効にする、またはその逆)。 私たちは、より安定した構造と教授法を見つけるために取り組んでいます。