グローバル最適化の問題
通常、最適化問題は次のように定式化されます。
与えられた関数f ( x )について、 xのすべての可能な値の中から、 f (x ')が最小(または最大)値を取るような値x'を見つけます。 さらに、 xは自然数、実数、複素数、ベクトルまたは行列などのさまざまなセットに属することができます。
関数f ( x )の極値とは、関数f ( x )の導関数がゼロに等しい点x 'を意味します。
関数の極値を見つけることが保証されている多くのアルゴリズムがあります。これは、与えられた開始点x 0の近くの局所的な最小値または最大値です。 このようなアルゴリズムには、たとえば、勾配降下アルゴリズムが含まれます。 ただし、実際には、1つのグローバルな極値に加えて、 xの特定の範囲で多くの局所的な極値を持つ関数のグローバルな最小値(または最大値)を見つけることがしばしば必要です。
勾配アルゴリズムはそのような関数の最適化に対処できません。なぜなら、それらの解は開始点近くの最も近い極値に収束するからです。 グローバルな最大値または最小値を見つける問題については、いわゆるグローバル最適化アルゴリズムが使用されます。 これらのアルゴリズムは、グローバルな極値を見つけることを保証しません。 は確率的アルゴリズムですが、特定のタスクに対してさまざまなアルゴリズムを試して、できれば最短時間でグローバル極値を見つける最大確率で、どのアルゴリズムが最適化にうまく対処するかを確認する以外に何もありません。
最も有名な(そして実装が難しい)アルゴリズムの1つは、遺伝的アルゴリズムです。
遺伝的アルゴリズムの一般的なスキーム
遺伝的アルゴリズムのアイデアは次第に生まれ、1960年代後半から1970年代初頭に形成されました。 遺伝的アルゴリズムは、John Hollandの本「Adaptation in Natural and Artificial Systems」(1975年)のリリース後に強力な発展を遂げました。
遺伝的アルゴリズムは、多数の世代にわたる個体群のモデリングに基づいています。 遺伝的アルゴリズムのプロセスでは、より良いパラメーターを持つ新しい個人が現れ、最も成功しない個人が死にます。 明確にするために、以下は遺伝的アルゴリズムで使用される用語です。
- 個体は、与えられた点xの目的関数の値とともに、可能な値のセットの中のxの 1つの値です。
- 染色体-xの値。
- 染色体-xがベクトルの場合のx iの値。
- フィットネス関数(フィットネス関数、目的関数)は最適化された関数f ( x )です。
- 母集団は、個人の集合です。
- 生成-遺伝的アルゴリズムの反復数。
各個人は、可能なすべてのソリューションのセットの中でxの 1つの値を表します。 最適化された関数の値(将来、簡潔にするために、関数の最小値を探していると仮定します)は、 xの各値に対して計算されます。 目的関数の重要度が低いほど、このソリューションは優れていると想定します。
遺伝的アルゴリズムは多くの分野で使用されています。 たとえば、ニューラルネットワークで重みを選択するために使用できます。 多くのCADシステムは、遺伝的アルゴリズムを使用して、指定された要件を満たすようにデバイスパラメーターを最適化します。 また、グローバル最適化アルゴリズムを使用して、ボード上の導体と要素の位置を選択できます。
遺伝的アルゴリズムの構造図を次の図に示します。
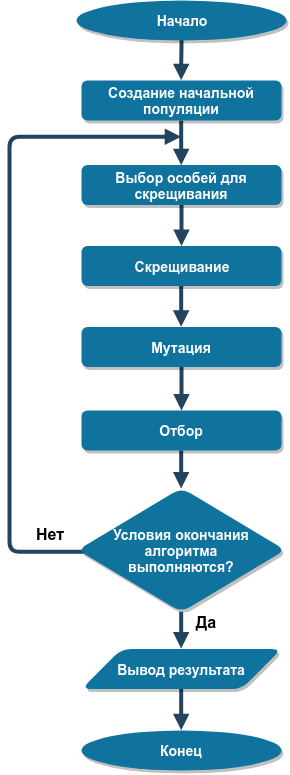
アルゴリズムの各段階をより詳細に検討します。
初期集団を作成する
アルゴリズムの最初の段階は、初期集団の作成、つまり、染色体xの値が異なる多数の個人の作成です。 原則として、初期集団はランダムな染色体値を持つ個人から作成されますが、グローバル極値がどこにあるかについての仮定がない限り、集団の染色体値がソリューション検索領域全体を比較的均一にカバーするようにします。
染色体をランダムに分散する代わりに、染色体を作成して、アルゴリズムのこの段階で作成される個人の数に応じて、特定のステップで初期x値が検索領域全体に均等に分散されるようにすることができます。
この段階でより多くの個体が作成されるほど、アルゴリズムが適切な解を見つける可能性が高くなり、また初期母集団のサイズが大きくなると、原則として、遺伝的アルゴリズムの反復(世代数)が少なくなります。 ただし、人口規模の増加に伴い、目的関数の計算数の増加と、アルゴリズムの後続の段階での他の遺伝子操作のパフォーマンスが必要になります。 つまり、人口規模が大きくなると、1世代の計算時間が長くなります。
原則として、集団のサイズは、遺伝的アルゴリズムの全作業を通じて一定である必要はありません。多くの場合、世代の数が増えるにつれて、集団のサイズを小さくすることができます。 時間が経つにつれて、増加する数の個人が希望するソリューションの近くに配置されます。 ただし、多くの場合、母集団のサイズはほぼ一定に維持されます。
交配のための個体の選択
母集団を作成した後、どの個体が交差操作に参加するかを決定する必要があります(次の段落を参照)。 この段階にはさまざまなアルゴリズムがあります。 最も簡単な方法は、各個人をそれぞれに交差させることですが、次のステップでは、目的関数の値を交差させて計算する操作を非常に多く実行する必要があります。
(目的関数の最小値を持つ)より成功した染色体を持つ個体と、子孫を残す能力を奪う目的関数がより多い(より悪い)個体と交配するより大きな機会を与える方が良いです。
したがって、この段階では、次の段階で交差操作を実行するために、個人のペア(または、より多くの個人が交差に参加できる場合は必ずしもペアではない)を作成する必要があります。
ここでは、さまざまなアルゴリズムを適用することもできます。 たとえば、ペアをランダムに作成します。 または、目的関数の値で昇順で個人を並べ替え、並べ替えられたリストの先頭に近い個人のペアを作成できます(たとえば、リストの前半、リストの最初の3分の個人など)。この方法は時間がかかるという点で悪いです。個人を分類します。
トーナメント方式がよく使用されます。 交差の各候補の役割に対して複数の個人がランダムに選択され、その中で目的関数の最高値を持つ個人が将来のペアに送信されます。 そして、ここでもランダム性の要素を導入することができ、目的関数の最悪の値を持つ個人が、目的関数の最高の値を持つ個人を「倒す」チャンスをわずかに与えます。 これにより、より不均一な集団を維持し、変性から保護することができます。 すべての個体がほぼ等しい染色体値を持っているという状況から。これは、アルゴリズムを極端な、おそらくグローバルではない状態に失速させることに等しい。
この操作の結果は、交差するパートナーのリストになります。
交配
交配は、新しい染色体値を持つ新しい個人を作成する遺伝子操作です。 新しい染色体は、親の染色体に基づいて作成されます。 ほとんどの場合、1組のパートナーを横断するプロセスで1人の娘が作成されますが、理論的には、より多くの子を作成できます。
クロスオーバーアルゴリズムは、さまざまな方法で実装することもできます。 染色体の種類が個人の数である場合、実行できる最も簡単なことは、親の染色体の算術平均または幾何平均として新しい染色体を作成することです。 多くのタスクではこれで十分ですが、ほとんどの場合、ビット演算に基づく他のアルゴリズムが使用されます。
ビットワイズ交配は、次の図に示すように、娘染色体が一方の親のビットの一部と他方の親のビットの一部で構成されるように機能します。
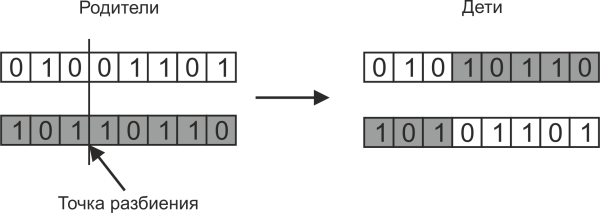
通常、親の分割ポイントはランダムに選択されます。 このような十字架を持つ2人の子供を作成する必要はありません。多くの場合、そのうちの1人に限定されます。
親染色体のスプリットポイントが1つの場合、そのような交差はシングルポイントと呼ばれます。 ただし、次の図に示すように、親の染色体がいくつかのセクションに分割されている場合は、マルチポイント分割を使用できます。
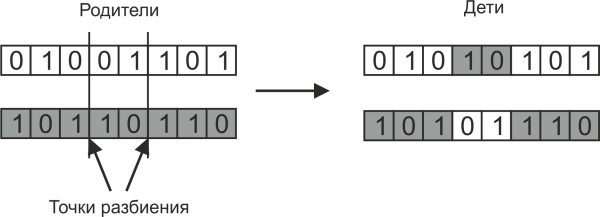
この場合、娘染色体のより多くの組み合わせが可能です。
染色体が浮動小数点数である場合、上記のすべての方法をそれらに適用できますが、それらはあまり効果的ではありません。 たとえば、前に説明したように、浮動小数点数がビットごとに交差する場合、多くの交差操作により、娘の染色体が作成されます。 倍精度浮動小数点数を使用すると、状況は特に悪化します。
この問題を解決するには、IEEE 754標準に準拠した浮動小数点数が3つの整数として保存されることを思い出す必要があります:S(1ビット)、MおよびN、そこから浮動小数点数はx =(-1) S × M×2 N 浮動小数点数を交差させる1つの方法は、最初に整数をコンポーネントS、M、Nに分割し、次に上記のビットごとの交差演算を数値MとNに適用し、次のいずれかの符号Sを選択することです親、および取得した値から娘染色体を作成します。
多くのタスクでは、個人は1つではなく複数の染色体を持ち、異なるタイプ(整数と浮動小数点数)になることさえあります。 次に、そのような染色体を横断するためのさらに多くのオプションがあります。 たとえば、娘の個人を作成する場合、親のすべての染色体を横断することも、変更せずに親の1つからいくつかの染色体を完全に転送することもできます。
突然変異
突然変異は、遺伝的アルゴリズムの重要な段階であり、個人の染色体の多様性をサポートするため、解がグローバルな最小値ではなく、あるローカル最小値にすぐに収束する可能性が低くなります。 突然変異は、交配によって作成されたばかりの個体の染色体のランダムな変化です。
原則として、突然変異の確率はそれほど高く設定されていないため、突然変異はアルゴリズムの収束を妨げません。そうでなければ、成功した染色体を持つ個人を台無しにします。
交差だけでなく、さまざまなアルゴリズムを突然変異に使用できます。 たとえば、1つの染色体または複数の染色体をランダムな値に置き換えることができます。 しかし、次の図に示すように、1ビットまたは複数ビットが染色体(または複数の染色体)で反転している場合、ほとんどの場合、ビット単位の突然変異が使用されます。
1ビットの突然変異:

いくつかのビットの突然変異:

突然変異のビット数は、事前定義するか、ランダム変数にすることができます。
突然変異の結果、個人はより成功し、より成功しなくなることがありますが、この操作により、親の個人が非ゼロの確率で出現する必要のないゼロと1のセットを持つ成功した染色体が可能になります。
セレクション
交配とその後の突然変異の結果、新しい個体が現れます。 選択段階がない場合、個人の数は指数関数的に増加し、新しい世代の人口ごとにますます多くのRAMと処理時間が必要になります。 したがって、新しい個人が登場した後、最も成功していない個人の人口をクリアする必要があります。 これが選択段階で発生することです。
選択アルゴリズムも異なる場合があります。 多くの場合、染色体がソリューション検索の特定の間隔内にない個人は、最初に破棄されます。
その後、一定の人口規模を維持するために、できるだけ成功していない個人をできるだけ多く落とすことができます。 選択のさまざまな確率的基準も適用できます。たとえば、個人の選択の確率は目的関数の値に依存する場合があります。
アルゴリズムの終了条件
遺伝的アルゴリズムの他の段階と同様に、アルゴリズムを終了するための基準がいくつかあります。
アルゴリズムを終了するための最も簡単な基準は、指定された反復(生成)数の後にアルゴリズムを中止することです。 ただし、遺伝的アルゴリズムは確率的であり、アルゴリズムの異なる開始が異なる速度で収束する可能性があるため、この基準は慎重に使用する必要があります。 通常、反復回数による終了基準は、アルゴリズムが多数の反復中に解決策を見つけることができない場合の追加の基準として使用されます。 したがって、十分な数を反復回数のしきい値として設定する必要があります。
もう1つの停止基準は、指定された反復回数で新しい最適なソリューションが表示されない場合、アルゴリズムが中断されることです。 これは、アルゴリズムがグローバルな極値を発見したか、ローカルな極値に留まっていることを意味します。
また、すべての個人の染色体が同じ意味を持つ場合、好ましくない状況があります。 これは、いわゆる縮退人口です。 この場合、ほとんどの場合、アルゴリズムは極端な状態に陥っており、必ずしもグローバルではありません。 成功した突然変異のみがこの状態から集団を抜け出すことができますが、突然変異の確率は通常小さなものによって確立され、突然変異がより成功した個人を作成するという事実からはほど遠いため、退化した集団の状況は通常、遺伝的アルゴリズムを停止する理由と見なされます。 この基準を確認するには、すべての個人の染色体を比較する必要があります。違いがあるかどうか、違いがない場合はアルゴリズムを停止します。
一部の問題では、グローバルな最小値を見つける必要はありませんが、目的関数の値が特定の値よりも小さい解決策を見つける必要があります。 この場合、次の反復で見つかった解がこの基準を満たす場合、アルゴリズムを事前に停止できます。
optlib
Optlibは、機能を最適化するために設計されたRust言語用のライブラリです。 これらの行を書いている時点では、遺伝的アルゴリズムのみがライブラリに実装されていますが、将来は新しい最適化アルゴリズムを追加してこのライブラリを拡張する予定です。 Optlibは完全にRustで書かれています。
Optlibは、MITライセンスの下で配布されるオープンソースライブラリです。
- githubのページ-https ://github.com/Jenyay/rust-optimization
- crates.ioのページ-https : //crates.io/crates/optlib
- ドキュメント-https://docs.rs/optlib
optlib ::遺伝的
遺伝的アルゴリズムの説明から、アルゴリズムの多くの段階をさまざまな方法で実装し、特定の目的関数に対してアルゴリズムが最短時間で正しい解に収束するようにそれらを選択できることがわかります。
optlib ::遺伝的モジュールは、「キューブから」遺伝的アルゴリズムを組み立てることができるように設計されています。 遺伝的アルゴリズムの作業が行われる構造のインスタンスを作成する場合、母集団の作成に使用するアルゴリズム、パートナーの選択、交配、突然変異、選択、およびアルゴリズムの中断に使用する基準を指定する必要があります。
ライブラリには、遺伝的アルゴリズムの段階で最も頻繁に使用されるアルゴリズムが既にありますが、対応するアルゴリズムを実装する独自のタイプを作成できます。
ライブラリでは、染色体が分数のベクトルである場合、つまり 関数f ( x )が最小化されている場合、 xは浮動小数点数のベクトル( f32またはf64 )です。
optlibを使用した最適化の例::遺伝
遺伝モジュールの詳細な説明を始める前に、Schwefel関数を最小化するための使用例を検討してください。 この多次元関数は次のように計算されます。
間隔-500 <= x i <= 500のSchweffel関数の最小値は、点x 'にあります。ここで、i = 1、2、...、Nのすべてのx i = 420.9687であり、この点での関数の値はf( x' )です。 = 0。
N = 2の場合、この関数の3次元画像は次のようになります。

この関数のレベルラインを作成すると、極値がより明確に表示されます。

次の例は、遺伝モジュールを使用して最小シュウェフェル関数を見つける方法を示しています。 この例は、examples / genetic-schwefelフォルダーのソースコードにあります。
//! . //! //! y = f(x), x = (x0, x1, ..., xi,... xn). //! x' = (420.9687, 420.9687, ...) //! xi - [-500.0; 500.0]. //! f(x') = 0 //! //! # //! * ` ` - . y = f(x). //! * `` - , x = (x0, x1, x2, ..., xn). //! * `` - x . //! * `` - . //! * `` - . use optlib::genetic; use optlib::genetic::creation; use optlib::genetic::cross; use optlib::genetic::goal; use optlib::genetic::logging; use optlib::genetic::mutation; use optlib::genetic::pairing; use optlib::genetic::pre_birth; use optlib::genetic::selection; use optlib::genetic::stopchecker; use optlib::testfunctions; use optlib::Optimizer; /// type Gene = f32; /// type Chromosomes = Vec<Gene>; fn main() { // // . xi [-500.0; 500.0] let minval: Gene = -500.0; let maxval: Gene = 500.0; // let population_size = 500; // xi . // 15- let chromo_count = 15; let intervals = vec![(minval, maxval); chromo_count]; // ( ) // optlib::testfunctions let goal = goal::GoalFromFunction::new(testfunctions::schwefel); // . // RandomCreator . let creator = creation::vec_float::RandomCreator::new(population_size, intervals.clone()); // // . let partners_count = 2; let families_count = population_size / 2; let rounds_count = 5; let pairing = pairing::Tournament::new(partners_count, families_count, rounds_count); // // , // - let single_cross = cross::FloatCrossExp::new(); let cross = cross::VecCrossAllGenes::new(Box::new(single_cross)); // // ( ). let mutation_probability = 15.0; let mutation_gene_count = 3; let single_mutation = mutation::BitwiseMutation::new(mutation_gene_count); let mutation = mutation::VecMutation::new(mutation_probability, Box::new(single_mutation)); // . , // . let pre_births: Vec<Box<genetic::PreBirth<Chromosomes>>> = vec![Box::new( pre_birth::vec_float::CheckChromoInterval::new(intervals.clone()), )]; // // , 1e-4 // 3000 (). let stop_checker = stopchecker::CompositeAny::new(vec![ Box::new(stopchecker::Threshold::new(1e-4)), Box::new(stopchecker::MaxIterations::new(3000)), ]); // . - . // NaN Inf. // , . let selections: Vec<Box<dyn genetic::Selection<Chromosomes>>> = vec![ Box::new(selection::KillFitnessNaN::new()), Box::new(selection::LimitPopulation::new(population_size)), ]; // . // , // . let loggers: Vec<Box<genetic::Logger<Chromosomes>>> = vec![ Box::new(logging::StdoutResultOnlyLogger::new(15)), Box::new(logging::TimeStdoutLogger::new()), ]; // let mut optimizer = genetic::GeneticOptimizer::new( Box::new(goal), Box::new(stop_checker), Box::new(creator), Box::new(pairing), Box::new(cross), Box::new(mutation), selections, pre_births, loggers, ); // optimizer.find_min(); }
この例を実行するには、ソースルートから次のコマンドを実行します。
cargo run --bin genetic-schwefel --release
結果は次のようになります。
Solution: 420.962615966796875 420.940093994140625 420.995391845703125 420.968505859375000 420.950866699218750 421.003784179687500 421.001281738281250 421.300537109375000 421.001708984375000 421.012603759765625 420.880493164062500 420.925079345703125 420.967559814453125 420.999237060546875 421.011505126953125 Goal: 0.015625000000000 Iterations count: 3000 Time elapsed: 2617 ms
サンプルコードのほとんどは、遺伝的アルゴリズムのさまざまな段階の特性オブジェクトの作成に関係しています。 記事の冒頭で書いたように、遺伝的アルゴリズムの各段階はさまざまな方法で実装できます。 optlib ::遺伝モジュールを使用するには、何らかの方法で各ステップを実装する特性オブジェクトを作成する必要があります。 次に、これらのオブジェクトはすべて、遺伝的アルゴリズムが実装されているGeneticOptimizer構造のコンストラクターに転送されます。
GeneticOptimizer構造体のfind_min()メソッドは、遺伝的アルゴリズムの実行を開始します。
基本的なタイプとオブジェクト
optlibモジュールの基本的な特徴
optlibライブラリは、プログラムが1つの最適化アルゴリズムから別の最適化アルゴリズムに簡単に切り替えられるように、将来、新しい最適化アルゴリズムを追加することを目的として開発されました。したがって、optlibモジュールには、単一の方法を含むOptimizer特性が含まれます- find_min()、実行時に最適化アルゴリズムを実行します。 ここで、タイプTはオブジェクトのタイプであり、ソリューションサーチスペース内のポイントです。 たとえば、上記の例では、これはVec <Gene>です(Geneはf32のエイリアスです)。 つまり、これは目的関数の入力にオブジェクトが渡される型です。
オプティマイザーの特性は、optlibモジュールで次のように宣言されます。
pub trait Optimizer<T> { fn find_min(&mut self) -> Option<(&T, f64)>; }
Optimizerトレイトのfind_min()メソッドは、Option <(&T、f64)>型のオブジェクトを返す必要があります。返されるタプルの&Tは見つかった解、f64は見つかった解の目的関数の値です。 アルゴリズムが解を見つけられなかった場合、find_min()関数はNoneを返すべきです。
多くの確率的最適化アルゴリズムはいわゆるエージェントを使用するため(遺伝的アルゴリズムの観点から、エージェントは個人です)、optlibモジュールには、単一のget_agents()メソッドを持つAlgorithmWithAgentsトレイトも含まれ、エージェントベクトルを返します。
次に、エージェントは、optlibからの別の特性を実装する構造です-Agent。
特性AlgorithmWithAgentsおよびAgentは、optlibモジュールで次のように宣言されます。
pub trait AlgorithmWithAgents<T> { type Agent: Agent<T>; fn get_agents(&self) -> Vec<&Self::Agent>; } pub trait Agent<T> { fn get_parameter(&self) -> &T; fn get_goal(&self) -> f64; }
AlgorithmWithAgentsとAgentの両方で、タイプTはオプティマイザーと同じ意味を持ちます。つまり、目的関数の値が計算されるオブジェクトのタイプです。
GeneticOptimizer構造-遺伝的アルゴリズムの実装
両方のタイプは、 GeneticOptimizer構造(OptimizerおよびAlgorithmWithAgents)に実装されています。
遺伝的アルゴリズムの各段階は、独自のタイプで表されます。これは、ゼロから実装するか、内部で利用可能なoptlib ::遺伝的実装のいずれかを使用できます。 遺伝的アルゴリズムの各段階では、optlib ::遺伝的モジュール内に、遺伝的アルゴリズムの段階で最も頻繁に使用されるアルゴリズムを実装する補助的な構造と機能を持つサブモジュールがあります。 これらのモジュールについては、以下で説明します。 また、これらのサブモジュールの中には、目的関数が浮動小数点数のベクトル(f32またはf64)から計算される場合のアルゴリズムのステップを実装するvec_floatサブモジュールがあります。
GeneticOptimizer構造のコンストラクターは、次のように宣言されます。
impl<T: Clone> GeneticOptimizer<T> { pub fn new( goal: Box<dyn Goal<T>>, stop_checker: Box<dyn StopChecker<T>>, creator: Box<dyn Creator<T>>, pairing: Box<dyn Pairing<T>>, cross: Box<dyn Cross<T>>, mutation: Box<dyn Mutation<T>>, selections: Vec<Box<dyn Selection<T>>>, pre_births: Vec<Box<dyn PreBirth<T>>>, loggers: Vec<Box<dyn Logger<T>>>, ) -> GeneticOptimizer<T> { ... } ... }
GeneticOptimizerのコンストラクターは、遺伝的アルゴリズムの各段階に影響を与える多くのタイプのオブジェクトと、結果の出力を受け入れます。
- ゴール:ボックス<dynゴール<T >>-目的関数。
- stop_checker:ボックス<dyn StopChecker <T >>-停止基準。
- 作成者:Box <dyn Creator <T >>-初期集団を作成します。
- ペアリング:ボックス<dynペアリング<T >>-交差するパートナーを選択するアルゴリズム。
- cross:Box <dyn Cross <T >>-交差アルゴリズム。
- 突然変異:ボックス<dyn突然変異<T >>-突然変異アルゴリズム。
- 選択:Vec <Box <dyn Selection <T> >>-選択アルゴリズムのベクトル。
- pre_births:Vec <Box <dyn PreBirth <T> >>-個人を作成する前に失敗した染色体を破壊するためのアルゴリズムのベクトル。
- ロガー:Vec <Box <dyn Logger <T> >>-遺伝的アルゴリズムのログを保存するオブジェクトのベクトル。
アルゴリズムを実行するには、Optimizerトレイトのfind_min()メソッドを実行する必要があります。 さらに、next_iterations()メソッドは、GeneticOptimizer構造体で使用できます。これは、find_min()メソッドの完了後に計算を続行するために使用できます。
アルゴリズムを停止した後、アルゴリズムのパラメーターまたは使用する方法を変更すると便利な場合があります。 これは、GeneticOptimizer構造体のメソッドreplace_pairing()、replace_cross()、replace_mutation()、replace_pre_birth()、replace_selection()、replace_stop_checker()を使用して実行できます。 これらのメソッドを使用すると、GeneticOptimizer構造体の作成時に渡される特性オブジェクトを置き換えることができます。
遺伝的アルゴリズムの主なタイプについては、次のセクションで説明します。
目標特性-目的関数
目標特性は、optlib ::遺伝モジュールで次のように宣言されます。
pub trait Goal<T> { fn get(&self, chromosomes: &T) -> f64; }
get()メソッドは、指定された染色体の目的関数の値を返す必要があります。
optlib ::遺伝::目標モジュール内には、Goal traitを実装するGoalFromFunction構造があります 。 この構造には、ターゲット関数である関数を受け取るコンストラクターがあります。 つまり、この構造を使用すると、通常の関数から目標タイプのオブジェクトを作成できます。
クリエイターの特性-初期集団の作成
作成者の特性は、初期集団の「作成者」を表します。 この特性は次のように宣言されます。
pub trait Creator<T: Clone> { fn create(&mut self) -> Vec<T>; }
create()メソッドは、初期集団の作成に基づいて染色体のベクトルを返す必要があります。
いくつかの浮動小数点数の関数が最適化される場合、 optlib ::遺伝::作成者:: vec_floatモジュールには、ランダムな方法で染色体の初期分布を作成するためのRandomCreator <G>構造があります。
以下、タイプ<G>は、染色体ベクター内の1つの染色体のタイプです(「染色体」という用語の代わりに「遺伝子」という用語が使用されることもあります)。 基本的に、染色体がタイプVec <f32>またはVec <f64>である場合、タイプ<G>はタイプf32またはf64を意味します。
RandomCreator <G>の構造は、次のように宣言されます。
pub struct RandomCreator<G: Clone + NumCast + PartialOrd> { ... }
ここで、NumCastはnum crateからのタイプです。 この型を使用すると、from()メソッドを使用して他の数値型から型を作成できます。
RandomCreator <G>構造を作成するには、次のように宣言されている新しい()関数を使用する必要があります。
pub fn new(population_size: usize, intervals: Vec<(G, G)>) -> RandomCreator<G> { ... }
ここで、population_sizeは母集団のサイズ(作成される染色体セットの数)、intervalは2つの要素のタプルのベクトルです-染色体セット内の対応する染色体(遺伝子)の最小値と最大値、およびこのベクトルのサイズは染色体セットに含まれる染色体の数を決定します各個人。
上記の例では、Schweffel関数はf32型の15個の変数に対して最適化されています。 各変数は、範囲[-500; 500]。 つまり、母集団を作成するには、間隔ベクトルに15タプル(-500.0f32、500.0f32)が含まれている必要があります。
タイプペアリング-交差するパートナーの選択
ペアリング特性は 、交配する個体を選択するために使用されます。 この特性は次のように宣言されます。
pub trait Pairing<T: Clone> { fn get_pairs(&mut self, population: &Population<T>) -> Vec<Vec<usize>>; }
get_pairs()メソッドは、母集団内のすべての個人を含むPopulation構造体へのポインターを取得します。また、この構造体を通じて、母集団内の最良の個人と最悪の個人を見つけることができます。
ペアリングget_pairs()メソッドは、交配する個体のインデックスを格納するベクトルを含むベクトルを返す必要があります。 交差アルゴリズム(次のセクションで説明します)に応じて、2人以上の個人が交差できます。
たとえば、インデックスが0と10、5と2、6と3の個体が交差する場合、get_pairs()メソッドはベクトルvec![Vec![0、10]、vec![5、2]、vec![ 6、3]]。
optlib ::遺伝::ペアリングモジュールには、さまざまなパートナー選択アルゴリズムを実装する2つの構造が含まれています。
RandomPairing構造の場合、Pairingタイプは、パートナーがランダムに選択されて交差するように実装されます。
トーナメント構造では、トーナメント方式が使用されるようにペアリング特性が実装されます。 トーナメント方式は、クロスに参加する各個人が別の個人を倒さなければならないことを意味します(勝った個人の目的関数の値はより小さくなければなりません)。 トーナメント方法が1ラウンドを使用する場合、これは2人の個人がランダムに選択され、これら2人の個人の中で目的関数の値が低い個人が将来の「家族」に入ることを意味します。 複数のラウンドが使用される場合、勝者は複数のランダムな個人に対して勝つ必要があります。
トーナメント構造コンストラクターは次のように宣言されます。
pub fn new(partners_count: usize, families_count: usize, rounds_count: usize) -> Tournament { ... }
ここに:
- Partners_count-横断に必要な個人の数。
- Families_count-「家族」の数、つまり 子孫を生成する個人のセット。
- rounds_count-トーナメントのラウンド数。
トーナメント方法のさらなる修正として、乱数生成器を使用して、目的関数の最悪の値を持つ個人を打ち負かすわずかな機会を与えることができます。 勝利の確率を目的関数の値に影響させ、2人の競合他社の差が大きいほど、最高の個人に勝つ可能性が高くなり、目的関数の値がほぼ等しい場合、1人の勝利の確率は50%近くになります。
タイプクロス-クロス
個体の「家族」が形成された後、交配のために、あなたは彼らの染色体を交配して、新しい染色体を持つ子供を得る必要があります。 交差ステージは、次のように宣言されるCrossタイプで表されます。
pub trait Cross<T: Clone> { fn cross(&mut self, parents: &[&T]) -> Vec<T>; }
cross()メソッドは、1セットの親を横断します。 このメソッドは、parentsパラメーター(親自身(個人ではなく)の染色体への参照からのスライス)を受け入れ、新しい染色体からベクターを返す必要があります。 親のサイズは、前のセクションで説明したペアリング特性を使用して、交配するパートナーを選択するアルゴリズムに依存します。 多くの場合、2人の親からの染色体に基づいて新しい染色体を作成するアルゴリズムが使用され、親のサイズは2に等しくなります。
optlib :: Genetic :: Crossモジュールには、Crossタイプがさまざまな交差アルゴリズムで実装されている構造が含まれています。
- VecCrossAllGenes-各個体が染色体のセットを持っているときに、染色体を横断するために設計された構造-これはベクターです。 VecCrossAllGenesコンストラクターは、親染色体ベクトルのすべての要素に適用されるCrossオブジェクトタイプを受け入れます。 この場合、親染色体ベクターのサイズは同じサイズでなければなりません。 上記の例では、使用される個人の染色体がVec <f32>型であるため、VecCrossAllGenes構造を使用しています。
- CrossMeanは、娘染色体として、親染色体の算術平均として計算される数があるように、2つの数を交差させる構造です。
- FloatCrossGeometricMeanは、娘染色体として親染色体の幾何平均として計算される数があるような方法で2つの数を交差させる構造です。 染色体は浮動小数点数でなければなりません。
- CrossBitwiseは、2つの数値の1ビットごとの1ポイント交差です。
- FloatCrossExp-浮動小数点数のビット単位の単一ポイント交差。 仮数と親の出展者が独立して交差します。 子供は両親の一人からサインを受け取ります。
optlib ::遺伝::クロスモジュールには、整数型と浮動小数点型のさまざまなタイプのビット単位の交差数の関数も含まれています。
タイプ突然変異-突然変異
交配後、染色体の多様性を維持するために得られた子供を変異させる必要があり、集団は縮退状態にスライドしませんでした。 母集団には、次のように宣言されている特性Mutationが意図されています。
pub trait Mutation<T: Clone> { fn mutation(&mut self, chromosomes: &T) -> T; }
Mutation形質の唯一のMutation()メソッドは、染色体への参照を取り、変更された可能性のある染色体を返す必要があります。 原則として、少数の突然変異の確率、たとえば数パーセントが確立されるため、親に基づいて取得された染色体は依然として保存され、人口が改善されます。
一部の突然変異アルゴリズムは、 optlib ::遺伝::突然変異モジュールに既に実装されています。
たとえば、このモジュールにはVecMutation構造が含まれています。これはVecCrossAllGenes構造と同様に機能します 。 染色体がベクターの場合、VecMutationはMutationオブジェクトタイプを受け入れ、ベクターのすべての要素に特定の確率で適用し、変異遺伝子(染色体ベクターの要素)を持つ新しいベクターを作成します。
optlib :: Genetic :: Mutationモジュールには、Mutation トレイトが実装されるBitwiseMutation構造も含まれています。 この構造は、送信された染色体内の指定された数のランダムビットを反転させます。 この構造をVecMutation構造と一緒に使用することをお勧めします。
出生前特性-出生前選択
交配と突然変異の後、通常は選択段階が発生し、最も失敗した個体が破壊されます。 ただし、optlib ::遺伝ライブラリでの遺伝的アルゴリズムの実装では、選択段階の前に、失敗した染色体を廃棄できる段階がもう1つあります。 個人の目的関数の計算には多くの時間がかかることが多く、染色体が既知の検索間隔に該当しない場合は計算する必要がないため、このステップが追加されました。 たとえば、上の例では、セグメントに該当しない染色体[-500; 500]。
このような事前フィルタリングを実行するために、 PreBirth特性は次のように宣言されています。
pub trait PreBirth<T: Clone> { fn pre_birth(&mut self, population: &Population<T>, new_chromosomes: &mut Vec<T>); }
PreBirth特性の唯一のpreBirth()メソッドは、前述のPopulation構造への参照と、new_chromosomes染色体ベクターへの可変参照です。 これは、突然変異段階の後に得られる染色体のベクターです。 pre_birth()メソッドを実装すると、明らかに問題の状態に適さない染色体がこのベクターから削除されます。
浮動小数点数のベクトルの機能が最適化されている場合、 optlib ::遺伝:: pre_birth :: vec_floatモジュールには、染色体が浮動小数点数のベクトルである場合に染色体を削除するように設計されたCheckChromoInterval構造と、ベクトルの要素が含まれます指定された間隔に該当しません。
CheckChromoInterval構造体のコンストラクターは次のとおりです。
pub fn new(intervals: Vec<(G, G)>) -> CheckChromoInterval<G> { ... }
ここで、コンストラクターは、2つの要素(染色体内の各要素の最小値と最大値)を持つタプルのベクトルを取ります。 したがって、間隔ベクトルのサイズは、個人の染色体ベクトルのサイズと一致する必要があります。 上記の例では、15タプル(-500.0f32、500.0f32)のベクトルが間隔として使用されました。
選択選択-選択
染色体の予備選択の後、個体が作成され、集団に配置されます( 集団構造)。 それぞれの個体を作成する過程で、目的関数の値が計算されます。 選択段階では、人口が無期限に増えないように、一定数の個体を破壊する必要があります。 このために、次のように宣言されている選択特性が使用されます。
pub trait Selection<T: Clone> { fn kill(&mut self, population: &mut Population<T>); }
kill()メソッドでSelectionトレイトを実装するオブジェクトは、破壊する必要がある母集団内の各個人に対して、 Individual構造(個々)のkill()メソッドを呼び出す必要があります。
母集団内のすべての個人をバイパスするには、Population構造体のIterMut()メソッドを返す反復子を使用できます。
多くの場合、いくつかの選択基準を適用する必要があるため、 GeneticOptimizer構造を作成するときに、コンストラクターはオブジェクトの選択タイプのベクトルを受け入れます。
遺伝的アルゴリズムの他のステージと同様に、 optlib ::遺伝的::選択モジュールはすでに選択ステージのいくつかの実装を提供しています。
- KillFitnessNaN-目的関数の値がNaNまたはInfである個人を破壊します。
- LimitPopulation-母集団のサイズが特定のサイズを超えないように、このような多数の最悪の個人を破壊します。
- optlib ::遺伝::選択:: vec_float :: CheckChromoInterval-浮動小数点数のベクトルの関数が最適化されている場合、染色体値が指定された間隔に収まらない個体を破壊します。 この構造は、遺伝的アルゴリズムの前の段階の構造optlib ::遺伝:: pre_birth :: vec_floatCheckChromoIntervalと同様に機能します。
StopChecker特性-停止基準
選択段階の後、遺伝的アルゴリズムを停止する時かどうかを確認する必要があります。 前述のように、この段階では、さまざまな停止アルゴリズムを使用することもできます。 StopChecker特性が実装されているアルゴリズムは、次のように宣言されており、アルゴリズムを中断します。
pub trait StopChecker<T: Clone> { fn can_stop(&mut self, population: &Population<T>) -> bool; }
ここで、アルゴリズムを停止できる場合、can_stop()メソッドはtrueを返す必要があります。そうでない場合、このメソッドはfalseを返す必要があります。
optlib :: Genetic :: Stopcheckerモジュールには、基本的な停止基準を持ついくつかの構造と、他の基準から組み合わせを作成するための2つの構造が含まれています。
- MaxIterations-世代番号による基準の破壊。 遺伝的アルゴリズムは、指定された回数の反復(世代)後に停止します。
- GoalNotChange-指定された反復回数内で新しいソリューションが見つからない場合にアルゴリズムが停止するブレーク基準。 または、言い換えれば、特定の世代数に対して、より成功した個人が現れない場合です。
- しきい値 -現時点での最適解の目的関数の値が指定されたしきい値よりも小さい場合に遺伝的アルゴリズムが中断される停止基準。
- CompositeAll — , ( - StopChecker). , - , .
- CompositeAny — , ( - StopChecker). , - , .
Logger —
Logger , , , . Logger :
pub trait Logger<T: Clone> { /// fn start(&mut self, _population: &Population<T>) {} /// , /// fn resume(&mut self, _population: &Population<T>) {} /// /// ( ) fn next_iteration(&mut self, _population: &Population<T>) {} /// fn finish(&mut self, _population: &Population<T>) {} }
optlib::genetic::logging , Logger:
- StdoutResultOnlyLogger — ;
- VerboseStdoutLogger — .
- TimeStdoutLogger — , .
GeneticOptimizer構造体のコンストラクターは、最後の引数として、Logger 特性 -objectsのベクトルを受け入れます。これにより、プログラムの出力を柔軟に構成できます。
最適化アルゴリズムをテストするための関数
シュヴェッフェル関数
最適化アルゴリズムをテストするために、多くの局所的な極値を持つ多くの機能が発明されました。モジュールoptlib :: testfunctionsは、アルゴリズムをテストすることができますいくつかの機能が含まれています。この記事の執筆時点では、optlib :: testfunctionsモジュールには2つの関数しか含まれていません。
これらの関数の1つはSchweffel関数で、これは記事の冒頭で説明しました。繰り返しますが、この関数は次のように書かれていることを思い出します。
x' = (420.9687, 420.9687, ...). .
optlib::testfunctions schwefel . N .
, , , , .
:
x' = (1.0, 2.0,… N). .
optlib::testfunctions paraboloid .
おわりに
optlib , . ( optlib::genetic ), , , , .
optlib::genetic. . , , , , .
, . , ( , ..)
さらに、最適化アルゴリズムをテストするために、(Schweffel関数に加えて)新しい分析関数を追加する予定です。
ここでも、optlibライブラリに関連付けられているリンクを思い出します。
- githubのページ-https ://github.com/Jenyay/rust-optimization
- crates.ioのページ-https : //crates.io/crates/optlib
- ドキュメント-https://docs.rs/optlib
コメント、追加、コメントをお待ちしております。