
/写真ミキヨシヒト CC
Habréのブログでは、IaaSの世界からの最新ニュースを定期的に共有しています。 たとえば、最近どのような変更がデータセンターを待っているかについて話しました。 また、大規模なインターネット企業がデータを保存する方法についても話しました。 今日、私たちは再びデータストレージのトピックに取り組み、有望な開発-DNAベースのストレージについて話したいと思います。
ハードドライブは、データストレージのために世界中のデータセンターで広く使用されており、耐久性で有名ではありません。 Backblazeチームは調査を実施し、HDDが情報を保存するのはわずか10年であることがわかりました。
残念ながら、これらは現代の現実です。ストレージデバイスは永遠に続くことはできません。 このため、世界中の研究者がデータを可能な限り長期間(理想的には無限に)保存する方法を模索しています。
そして、彼らは彼を見つけました。 すべての質問に対する答えはDNAにあると信じられています-それは高い記録密度(1 mm3あたり1エクサバイト)と耐久性(確立された減衰期間は500年以上です)を持っています。
デジタルユニバースのサイズは、2017年までに16ゼタバイトを超えます。 このデータの大部分はアーカイブに保存されます。 たとえば、Facebookは最近、1エクサバイトのデータのコールドストレージ用に別のデータセンターを構築しました。 同じ量の情報が1 mm3のDNAに収まります。
データをDNAに保存するには、デジタルデータをDNAヌクレオチドのシーケンスに変換する、DNA分子を合成する、直接データを保存するという3つの段階があります。 データを読み取るには、DNA分子から目的の配列を分離し、元の形式に変換する必要があります。
DNAストレージの操作には困難があること、たとえばデータ暗号化のコストについて疑問があることは注目に値しますが、研究者は医療技術が発展するにつれて減少すると確信しています。
これが起こることです。 合成とシーケンスを実行する時間は指数関数的に減少し、効率の増加はムーアの法則に従います 。
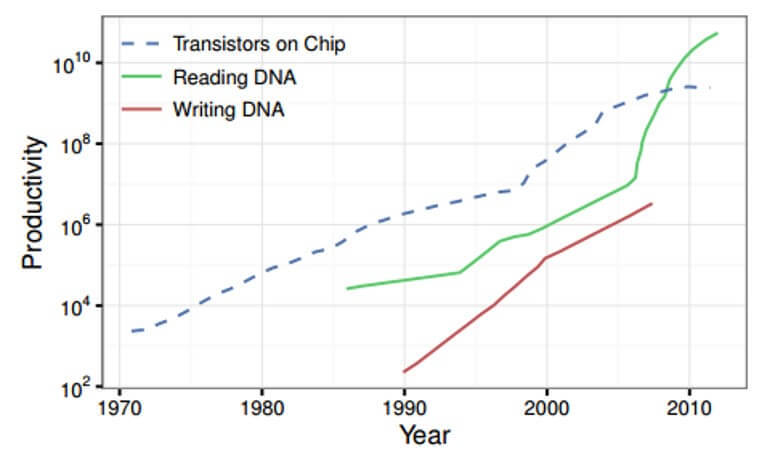
チップ上のトランジスタ数の増加と比較したDNA合成の傾向
さらに、このような手順のコストも下がります。 以前は、ヒトゲノムの解読コストは数百万ドルでしたが、今日では数百に下がっています。
これらの前向きな傾向により、ワシントン大学の科学者はDNAベースのキーバリューストレージシステムの開発を開始しました。 彼らはそのようなシステムを現代のアーキテクチャで使用する可能性を探求したいと考えています。
情報エンコーディングプロセス
DNAには、アデニン(A)、シトシン(C)、グアニン(G)、チミン(T)の4種類のヌクレオチドが含まれています。 DNA鎖は、これらのヌクレオチドの線形配列です。 したがって、4つのコードシンボル(A、C、G、およびT)があるため、バイナリデータを格納するための明白なアプローチは、4桁の数値システムでエンコードすることです。たとえば、0 = A、1 = C、2 = G、3 = T ただし、合成とシーケンスはエラーが発生しやすいことに留意してください。
次の図に示すように、バイナリ情報が4倍ではなく3進数でエンコードされている場合、エラーの可能性を減らすことができます。 ソースバイナリデータの3進数システムへの非効率的な変換を回避するために、ハフマンコードが使用されます。
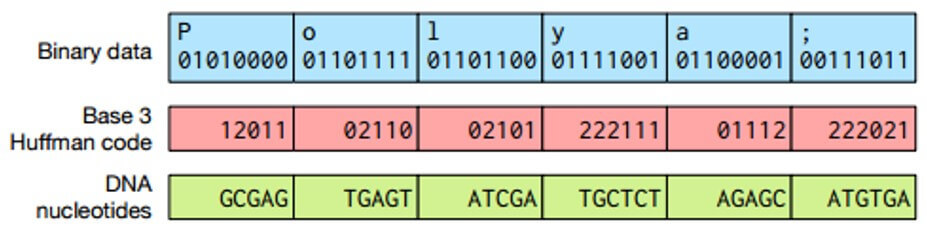
バイナリデータとDNAヌクレオチドの比較
3桁のそれぞれは、表(下記)に従ってDNAヌクレオチドに対応し、チェーン内のヌクレオチドは繰り返されないため、シーケンスエラーが減少します。
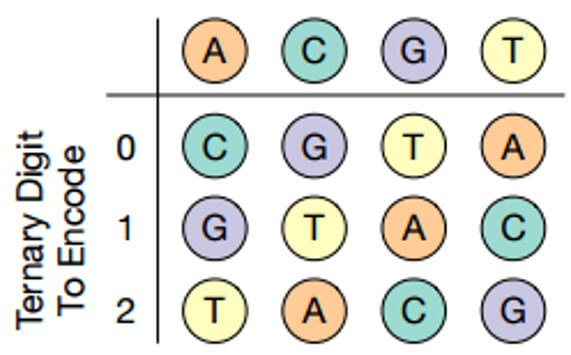
ヌクレオチドコード表
データへのランダムアクセスを許可するために、科学者はキーのユニークなプライマーシーケンスへの変換を整理しました。 プライマーは、増幅する必要がある領域の開始と終了を定義する短い合成ストランドです。
プライマーは、ポリメラーゼ連鎖反応を使用してランダムアクセスを提供します。これにより、溶液中のDNAのコピーが多数生成されます。 特定のオブジェクトのチェーンには共通のプライマーがあり、同じプライマーを持つ異なるチェーンのアドレスは異なります。
「ポリメラーゼ連鎖反応(PCR)のプライマーとして使用される配列を制御することにより、溶液中のどの鎖が増幅を受けるかを示すことができます。 ソリューションのキーの値を読み取るには、このキーに対応するプライマーを使用してPCRを実行するだけです」と科学者は言います。
DNAベースのストレージシステム
DNAベースのストレージシステムは、データをエンコードするDNAシンセサイザー、データストレージコンテナー、およびDNAシーケンスを読み取って「数字」に変換するDNAシーケンサーで構成されます。
短い形式でデータを読み書きするプロセスを下の図に示します。
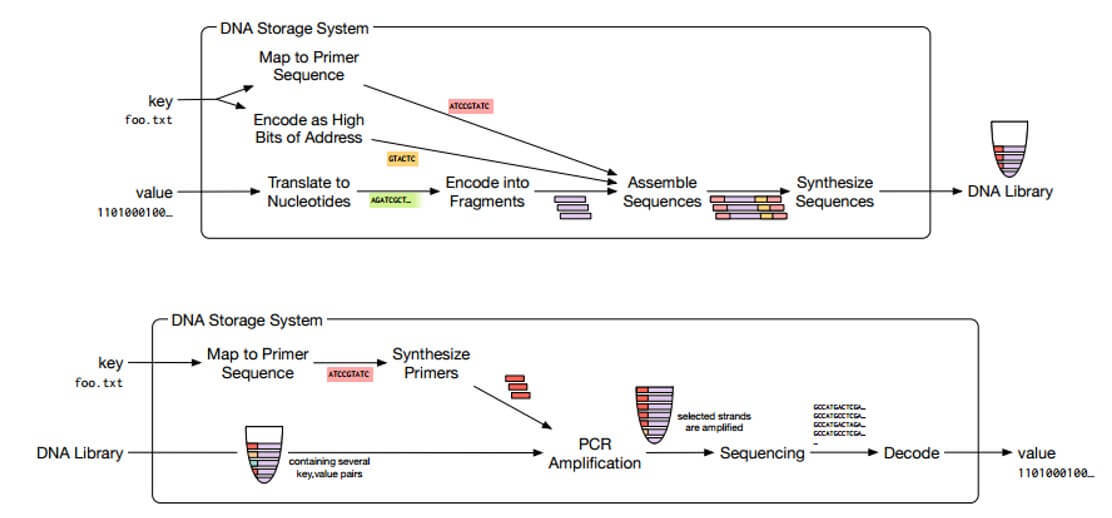
DNAストレージ
読み取り中に、DNAサンプルがプールから削除され、後続の操作に使用できるDNAの量が減少します。 しかし、DNAは簡単にコピーされるため、必要に応じて、プールは欠落しているフラグメントを簡単に埋めることができます。 連続増幅中に問題が発生した場合、プールで読み取った後、DNA合成を再度実行できます。
おわりに
将来的には、そのようなシステムにより、微視的なメディアに大量のデータを保存できるようになる可能性があります。 約100,000 PBのデータを保存できる、100 mm3の容量の「フラッシュドライブ」を想像してください。
ただし、これまでのところ、このような技術の導入に対する最大の障害は時間です。 DNA分子の解読と読み取りには何時間もかかります。 したがって、このタイプのストレージは、頻繁に使用されるデータのコンテンツにはほとんど適していませんが、データセンターの長期ストレージに関する理解をゆがめる可能性があります。
PSHabréのブログのテーマに関するその他の資料:
PPS IaaSプロバイダー1cloudに精通し、週末にその機能をテストする時間がある場合に備えて、実用的なガイドラインへのリンクを用意しました。