この記事では、ロジックの単純な変更、並列処理、
Rcpp
を含むいくつかのアプローチについて説明します。速度を数桁向上させることで、1億行以上のデータを処理できるようになります。
forループと条件ステートメント(if-else)を使用してコードを高速化し、データセット(データフレーム、df)に追加される列を作成してみましょう。 以下のコードは、この初期データセットを作成します。
# col1 <- runif (12^5, 0, 2) col2 <- rnorm (12^5, 0, 2) col3 <- rpois (12^5, 3) col4 <- rchisq (12^5, 2) df <- data.frame (col1, col2, col3, col4)
最初の部分では、ベクトル化、真の条件のみ、ifelse。
この部分では、適用、バイトコンパイル、Rcpp、data.table、結果。
which()を使用する
which()
コマンドを使用し
which()
行を選択すると、
Rcpp
速度の3分の1を達成できます。
# system.time({ want = which(rowSums(df) > 4) output = rep("less than 4", times = nrow(df)) output[want] = "greater than 4" })
# = 3 () user system elapsed 0.396 0.074 0.481
forループの代わりに適用関数ファミリーを使用します
apply()
関数を使用して同じロジックを実装し、ベクトル化されたforループと比較します。 結果は注文数の増加とともに増加しますが、
ifelse()
およびループ外でチェックが行われたバージョンよりも遅くなります。 これは便利かもしれませんが、複雑なビジネスロジックには多少の工夫が必要になる場合があります。
# apply system.time({ myfunc <- function(x) { if ((x['col1'] + x['col2'] + x['col3'] + x['col4']) > 4) { "greater_than_4" } else { "lesser_than_4" } } output <- apply(df[, c(1:4)], 1, FUN=myfunc) # 'myfunc' df$output <- output })

Rでの適用とforループの使用
関数自体の代わりにコンパイラパッケージのcmpfun()関数にバイトコンパイルを使用する
結果の時間は通常の形式よりもわずかに長いため、これはおそらくバイトコンパイルの効率を示す最良の例ではありません。 ただし、より複雑な関数の場合、バイトコンパイルが効果的であることが証明されています。 たまに試してみる価値があると思います。
# library(compiler) myFuncCmp <- cmpfun(myfunc) system.time({ output <- apply(df[, c (1:4)], 1, FUN=myFuncCmp) })

適用、forループおよびバイトコードのコンパイル
rcppを使用する
新しいレベルに到達しましょう。 これに先立ち、さまざまな戦略を使用して速度とパフォーマンスを向上させ、
ifelse()
を使用する
ifelse()
最も効率的であることがわかりました。 別のゼロを追加するとどうなりますか? 以下では、
Rcpp
で同じロジックを実装し、1億行のデータセットを使用します。
Rcpp
と
ifelse()
の速度を比較します。
library(Rcpp) sourceCpp("MyFunc.cpp") system.time (output <- myFunc(df)) # Rcpp
以下は、Rcppパッケージを使用してC ++で実装された同じロジックです。 以下のコードをRセッション作業ディレクトリに「MyFunc.cpp」として保存します(または、フルパスを使用してsourceCppを使用する必要があります)。 コメント
// [[Rcpp::export]]
必須であり、Rから実行する関数の直前に配置する必要があることに注意してください。
// MyFunc.cpp #include using namespace Rcpp; // [[Rcpp::export]] CharacterVector myFunc(DataFrame x) { NumericVector col1 = as(x["col1"]); NumericVector col2 = as(x["col2"]); NumericVector col3 = as(x["col3"]); NumericVector col4 = as(x["col4"]); int n = col1.size(); CharacterVector out(n); for (int i=0; i 4){ out[i] = "greater_than_4"; } else { out[i] = "lesser_than_4"; } } return out; }
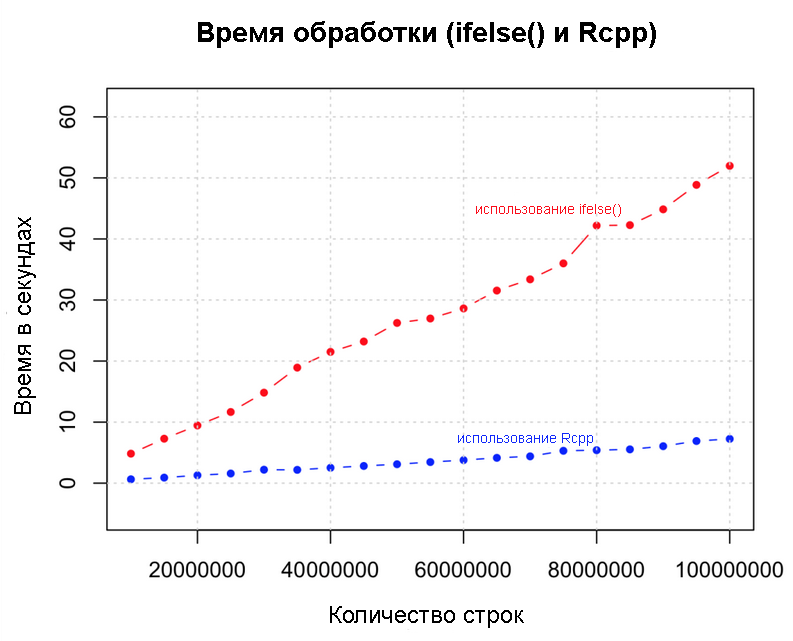
Rcpp
と
ifelse
パフォーマンス
マルチコアコンピューターを使用している場合は、並列処理を使用します
並列処理:
# library(foreach) library(doSNOW) cl <- makeCluster(4, type="SOCK") # for 4 cores machine registerDoSNOW (cl) condition <- (df$col1 + df$col2 + df$col3 + df$col4) > 4 # system.time({ output <- foreach(i = 1:nrow(df), .combine=c) %dopar% { if (condition[i]) { return("greater_than_4") } else { return("lesser_than_4") } } }) df$output <- output
変数を削除し、できるだけ早くメモリをクリアする
特に長いループの前に、できるだけ早く
rm()
を使用してコード内の不要なオブジェクトを削除します。 ループの各反復の終わりに
gc()
が役立つ場合があります。
より少ないメモリを使用するデータ構造を使用する
Data.table()
は、メモリをオーバーロードしないため、素晴らしい例です。 これにより、データフェデレーションなどの操作が高速化されます。
dt <- data.table(df) # data.table system.time({ for (i in 1:nrow (dt)) { if ((dt[i, col1] + dt[i, col2] + dt[i, col3] + dt[i, col4]) > 4) { dt[i, col5:="greater_than_4"] # 5- } else { dt[i, col5:="lesser_than_4"] # 5- } } })
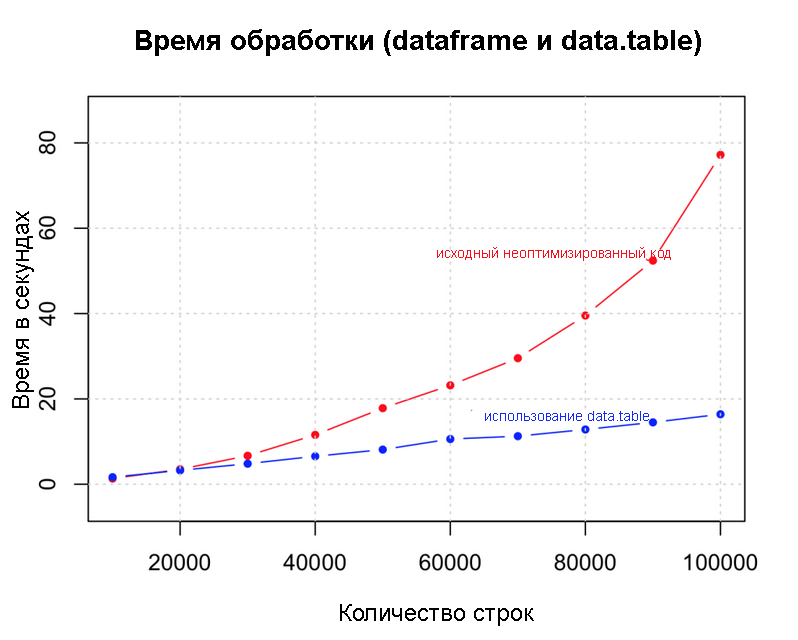
データフレームとdata.table
速度:結果
方法:速度、dfライン/経過時間= 1秒あたりnライン
オリジナル: 1X、120000 / 140.15 = 856.2255行/秒(1に正規化)
ベクトル化: 738X、120,000 / 0.19 = 631578.9行/秒
真の条件のみ: 1002X、120,000 / 0.14 = 857,142.9ライン/秒
ifelse: 1752X、1200000 / 0.78 = 1500000行/秒
これ: 8806X、2985984 / 0.396 = 7540364ライン/秒
Rcpp : 13476X、1200000 / 0.09 = 11538462ライン/秒
上記の数値は概算であり、ランダムな開始に基づいています。
data.table()
結果の計算、コードのバイトコンパイル、および並列化はありません。これらは、それぞれの場合と使用方法によって大きく異なるためです。