はじめに:ネットワークの可視化
ネットワークの視覚化を設計する際の主なことは、達成する必要がある目標です。 どの構造特性を強調したいですか?

ネットワークマップは、唯一のグラフ視覚化ツールとはほど遠い-場合によっては、ネットワークを表す他の形式、主要なプロパティの単純なグラフでさえ、より望ましいものです。

ネットワークマップでは、他の視覚化形式と同様に、最終結果に影響するいくつかの重要な設定があります。 主なものは、色、サイズ、形状、相対的な配置です。
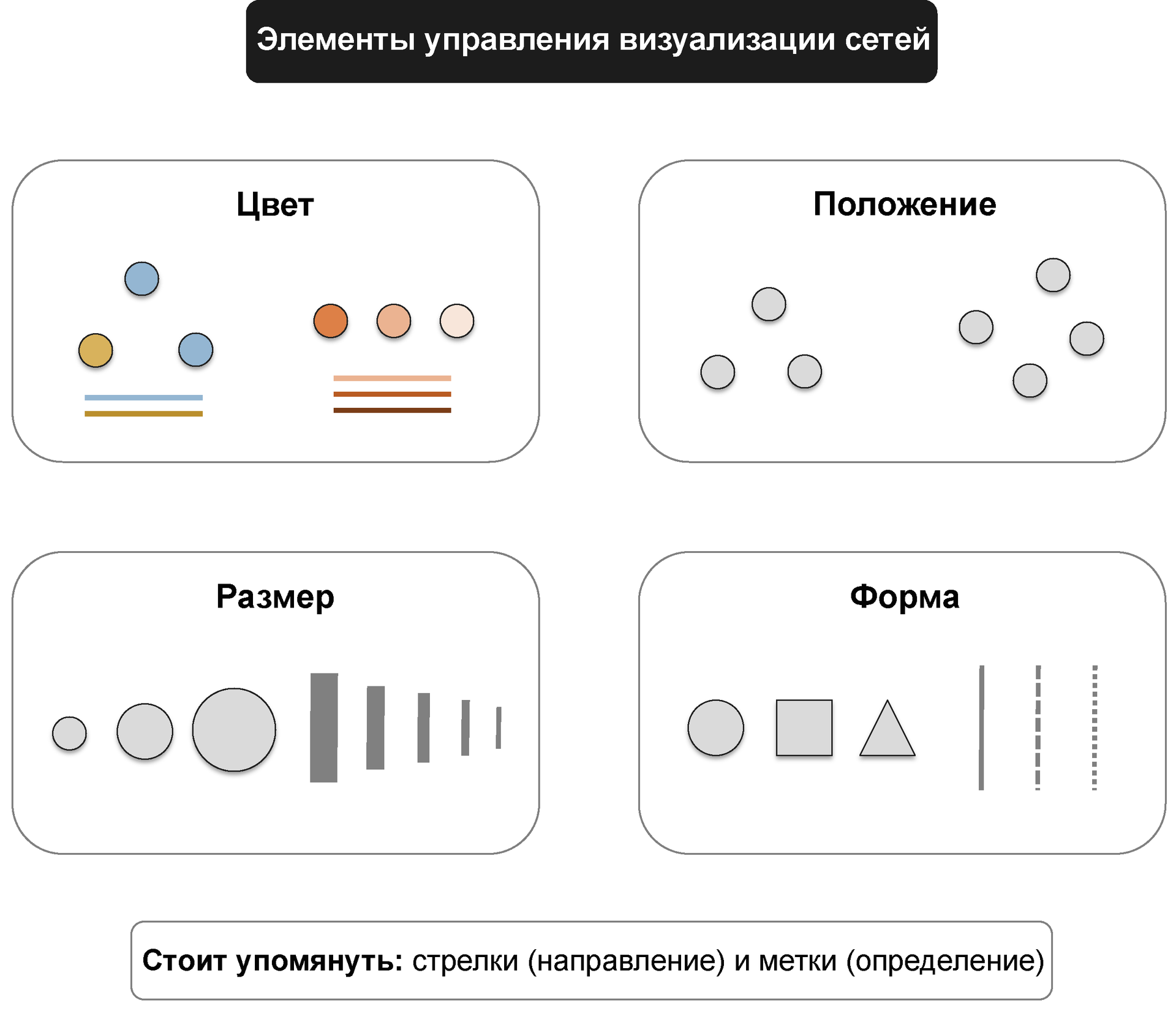
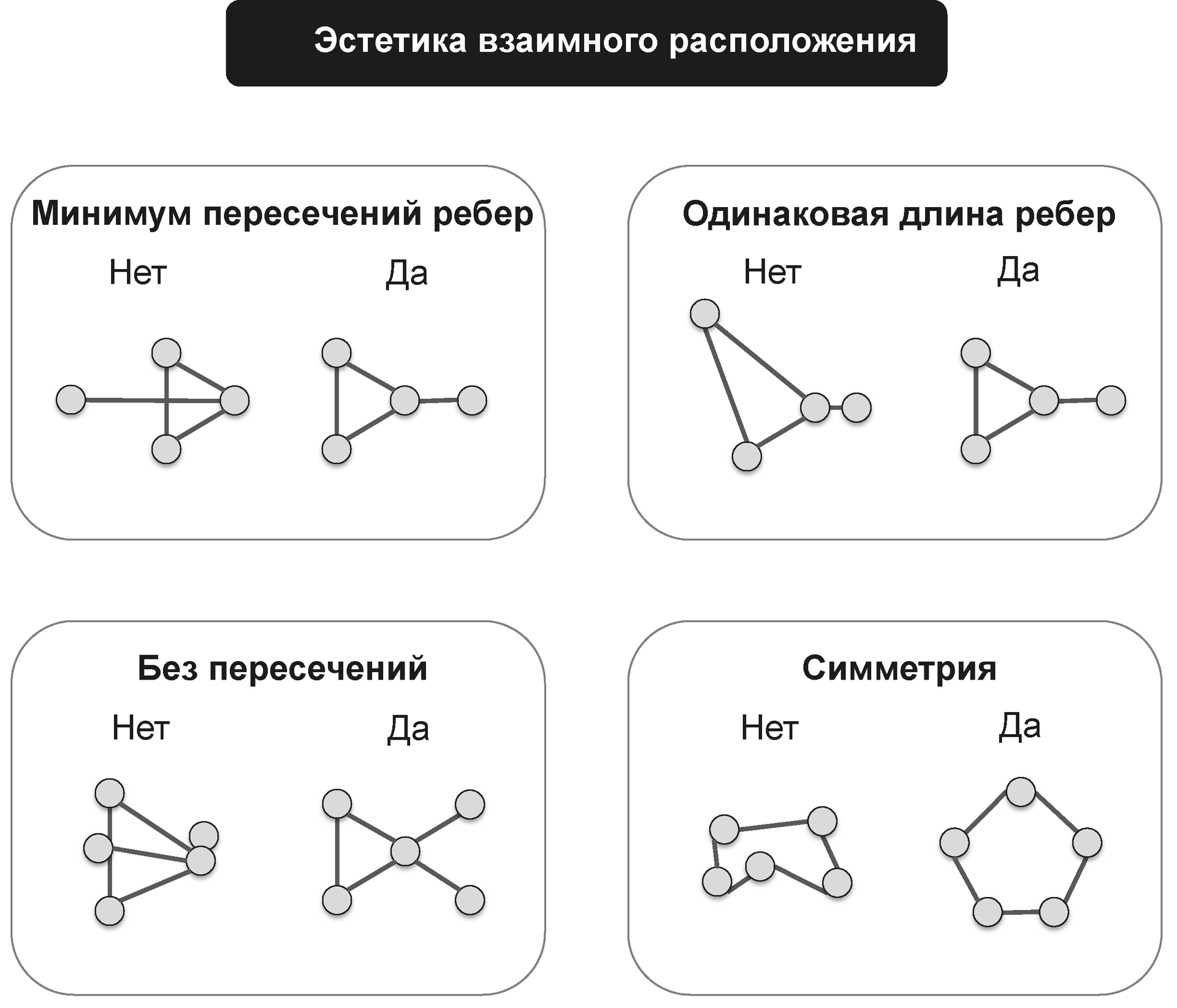
最新のグラフ表現は、パフォーマンス要件と審美的な考慮事項に基づいて最適化されます。 特に、グラフ内で同じ長さのエッジを確立するには、エッジのオーバーラップと交差を最小限に抑える必要があります。
データ形式、サイズ、準備
このガイドでは、主に2つの小さなデータセットを使用します。 どちらにもメディアに関する情報が含まれています。 1つには、ニュースリソースにハイパーリンクと言及のネットワークが含まれています。 もう1つは、オブジェクトとメディアの消費者との間のリンクのネットワークです。 例のデータは少ないですが、生成された視覚化の多くのアイデアは中規模および大規模ネットワークに拡張できます。 同じ理由で、たとえば頂点文字の形状など、視覚的な手段を使用することはめったにありません。大きなグラフでは区別することはほとんど不可能です。 さらに、非常に大規模なネットワークを表示する場合、頂点グループの識別と表示に集中する必要があるため、エッジを非表示にすることもできます。 一般的に、Rを使用して視覚化できるネットワークのサイズは、マシンのRAMの量によってのみ制限されます。 しかし、大規模なネットワークを巨大なポンポンの形で視覚化することは、多くの場合、主要なグラフプロパティを持つグラフよりもはるかに有用でないことを強調する必要があります。
このガイドでは、続行する前にインストールする必要のあるいくつかの主要なパッケージを使用しています。 さらにいくつかのライブラリが言及されますが、それらはオプションであり、スキップできます。 igraph ( Gabor TsardiとTamas Nepushがサポート )、 sna 、 network ( Carter ButtsとStatnetチームがサポート)、 ndtv ( Sky Bender deMollがサポート)のメインライブラリが使用されます。
install.packages("igraph") install.packages("network") install.packages("sna") install.packages("ndtv")
データセット1:エッジのリスト
動作する最初のデータセットは、「Media-Example-NODES.csv」と「Media-Example-EDGES.csv」の2つのファイルで構成されています( こちらからダウンロードしてください )。
nodes <- read.csv("Dataset1-Media-Example-NODES.csv", header=T, as.is=T) links <- read.csv("Dataset1-Media-Example-EDGES.csv", header=T, as.is=T)
データを調べます。
head(nodes) head(links) nrow(nodes); length(unique(nodes$id)) nrow(links); nrow(unique(links[,c("from", "to")]))
一意の「from」-「to」の組み合わせよりも多くのエッジがあることに注意してください。 つまり、データでは、2つの頂点間に複数の接続がある場合があります。 2つのノード間で同じタイプのすべてのエッジを折りたたみ、「from」、「to」、および「type」による
aggregate()
関数を使用して重みを合計します。
links <- aggregate(links[,3], links[,-3], sum) links <- links[order(links$from, links$to),] colnames(links)[4] <- "weight" rownames(links) <- NULL
データセット2:マトリックス
nodes2 <- read.csv("Dataset2-Media-User-Example-NODES.csv", header=T, as.is=T) links2 <- read.csv("Dataset2-Media-User-Example-EDGES.csv", header=T, row.names=1)
データを調べます。
head(nodes2) head(links2)
links2が双方向ネットワークのペアリングマトリックスであることを確認できます。
links2 <- as.matrix(links2) dim(links2) dim(nodes2)
ネットワークの視覚化: igraphの最初のステップ
ソースデータをigraphネットワークに変換することから始めましょう。 これを行うには、igraph graph.data.frame関数を使用します。この関数は、2つのデータブロックを入力として使用します:dと頂点。
- dは、ネットワークのエッジを示します。 最初の2列には、各エッジの開始頂点と終了頂点の識別子が含まれています。 次の列には、リブのパラメーター(重量、タイプ、ラベル、その他)があります。
- 頂点は、頂点識別子の列で始まります。 以下のすべての列は、頂点パラメーターとして解釈されます。
library(igraph) net <- graph.data.frame(links, nodes, directed=T) net
## IGRAPH DNW- 17 49 -- ## + attr: name (v/c), media (v/c), media.type (v/n), type.label ## (v/c), audience.size (v/n), type (e/c), weight (e/n)
igraphオブジェクトの説明は4文字で始まります。
- DまたはU-それぞれ、有向グラフまたは無向グラフの場合。
- N-名前付きグラフの場合(ノードには
name
属性があります)。 - W-重み付きグラフの場合(リンクには
weight
属性があります)。 - B-両面グラフの場合(ノードには
type
属性があります)。
次の2つの数値(17 49)は、グラフ内の頂点とエッジの数を示します。 この説明では、頂点とエッジのプロパティも示します。次に例を示します。
-
(g/c)
-グラフレベルの行レベルプロパティ -
(v/c)
-頂点レベルの行プロパティ -
(e/n)
-エッジレベルのプロパティ番号
また、頂点、エッジ、およびそれらの属性に簡単にアクセスできます。
E(net) # "net" V(net) # "net" E(net)$type # "type" V(net)$media # "media" # : net[1,] net[5,7]
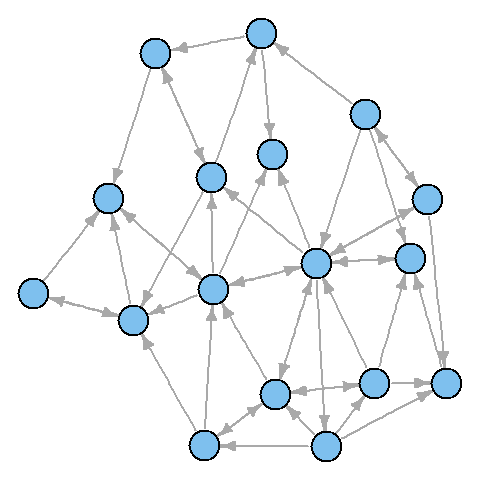
igraphネットワークができたので、最初にそれを構築することができます。
plot(net) # !

あまり美しくないことがわかりました。 グラフのループを削除して、画像の改善を始めましょう。
net <- simplify(net, remove.multiple = F, remove.loops = T)
あなたが
simplify
ようなコマンドでそれらの重みを合計することで複数のエッジを1つに折りたたむことができることに気づくことができます
simplify(net, edge.attr.comb=list(Weight="sum","ignore"))
。 問題は、結合する際にエッジのタイプが考慮されないことです(データでは「ハイパーリンク」-リンクと「言及」-参照)。
また、矢印のサイズを小さくして、ラベルを削除します(ラベルを
NA
設定して)。
plot(net, edge.arrow.size=.4,vertex.label=NA)
パート2:R.チャートのフォントと色