 この記事はR言語についてです 。 元ソ連ではMatlab、特にPythonほど広くはありませんが、確かに注目に値します。 Rは実際にはデータサイエンスの標準であることに注意する必要があります(ただし、 ここではデータサイエンティストだけがR であるとは書かれていません)。 豊富な構文、レガシーコードとの互換性(科学アプリケーションで非常に重要)、RStudioの便利な開発環境、CRANの膨大な数のライブラリの存在により、Rはそのようになります。
この記事はR言語についてです 。 元ソ連ではMatlab、特にPythonほど広くはありませんが、確かに注目に値します。 Rは実際にはデータサイエンスの標準であることに注意する必要があります(ただし、 ここではデータサイエンティストだけがR であるとは書かれていません)。 豊富な構文、レガシーコードとの互換性(科学アプリケーションで非常に重要)、RStudioの便利な開発環境、CRANの膨大な数のライブラリの存在により、Rはそのようになります。
しかし、いつものように、コインの裏(または、通訳R)は計算速度です。 Rはベクトル化された計算をサポートします— 関数の適用ファミリーはループの使用を避けます。 しかし、これは、一般的に、構文糖であり、多くの場合、* applyを使用することによる「加速」の効果は直接負です。 この背景に対して、計算の並列化は完全に論理的なステップのようです(低レベル言語への移行を考慮しない場合)。 この手順でコードを処理するのが難しいことは秘密ではありません。
さらに作業を進めるには、「foreach」(foreachコンストラクトを使用できるようにする)と「doSNOW」(並列コンピューティングのサポートを提供する)の2つのパッケージが必要です。 CRANから次のようにインストールできます。
install.packages(c("foreach","doSNOW"))
次のコマンド-操作のために3つのプロセッサコアの「クラスター」を準備します。
library(foreach) library(doSNOW) cluster <- makeCluster(3, type = "SOCK", outfile="") registerDoSNOW(cluster)
1から10000までの数の平方の合計が計算される例を考えてみましょう。
system.time ({ sum.par <- foreach(i=1:10000, .combine='+') %dopar% { i^2 }})
原則として、結果をグループ化する方法を指定する必要があることを除いて、すべてが通常のサイクルに非常に似ています-この場合、それは追加操作です(多くの場合、これらは関数c、rbind、cbind-または自分で作成した関数です)。 どの順序でグループ化するかが重要でない場合は、.inorder = F引数を使用すると、計算を多少高速化できます。 %dopar%の代わりに、%do%ディレクティブが使用される場合、計算は順番に実行されます(つまり、通常のループのように)。 ところで、このような単純なタスクの場合、シーケンシャルバージョンの実行時間は著しく短くなります。 並列バージョンでのデータ操作のオーバーヘッドコストは、貸借対照表の最後の場所から大きく離れています。
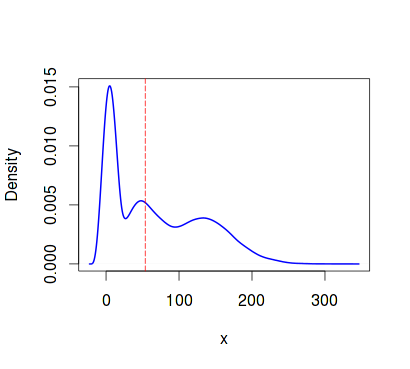
さらに難しいタスクに移りましょう。 ランダム変数xのベクトルがあるとします。
非表示のテキスト
trials <- 10000 n <- 24000 alfa <- 1.6 beta <- 3 set.seed(1) x <- c(rgamma(n/3, shape=alfa, scale=beta), rgamma(n/3, shape=3*alfa, scale=4*beta), rgamma(n/3, shape=10*alfa, scale=3*beta)) plot(density(x, kernel="cosine"),lwd=2, col="blue", xlab="x",ylab="Density", main="Density plot") abline(v = median(x), col = "red", lty=5)
信頼区間で中央値を推定します。 これを行うには、 ブートストラップを適用し、シリアルコードとパラレルの実行時間をそれぞれ測定します。
set.seed(1) seq.time <- system.time({ seq.medians <- foreach(icount(trials), .combine=cbind) %do% { median(sample(x, replace = T)) } })
> quantile(seq.medians, c(.025, .975)) 2.5% 97.5% 52.56311 54.99636 > seq.time user system elapsed 19.884 0.252 20.138
set.seed(1) par.time <- system.time({ par.medians <- foreach(icount(trials), .combine=cbind) %dopar% { median(sample(x, replace = T)) } })
> quantile(par.medians, c(.025, .975)) 2.5% 97.5% 52.56257 54.94890 > par.time user system elapsed 4.252 0.420 10.893
違いは非常に顕著です。 同じタスクの順次「ベクトル化」バージョンの実行時間を評価してみましょう(適用機能を使用)。
set.seed(1) seq.time.ap <- system.time({ seq.medians.ap <- apply(matrix(sample(x, trials*n, replace = T), trials, n), 1, median) })
> quantile(seq.medians.ap, c(.025, .975)) 2.5% 97.5% 52.56284 54.98266 > seq.time.ap user system elapsed 23.732 1.728 25.472
同様のアプローチを使用して、機械学習アルゴリズムのパラメーターを決定できます。 たとえば、RSNNSパッケージにはElmanネットワークをトレーニングする機能があります (私が知っている限り、Rでは一般に、リカレントネットワークを操作する他のパッケージはありません)。 ニューロンの数と学習率の選択はかなりリソースを消費するタスクであるため、並列化するのが理にかなっています。
球面の例から、より差し迫った問題に移ります。 これを行うには、2つのパッケージをインストールします。
install.packages(c("ElemStatLearn","RSNNS"))
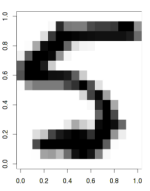
ElemStatLearnパッケージから、zip.trainとzip.testの2つのデータセットが必要です。これらには、gzip圧縮された手書き数字画像が含まれています。 データセットは非常に膨大であるため、それらの一部のみを取り上げましょう。それぞれ、ネットワークトレーニング、検証、テスト用です。
非表示のテキスト
library("ElemStatLearn") library(RSNNS) data(zip.train) data(zip.test) image(zip2image(zip.train, 1000), col=gray(255:0/255)) train <- zip.train[1:1000,-1] trainC <- decodeClassLabels(zip.train[1:1000,1]) valid <- zip.train[1001:1200,-1] validC <- zip.train[1001:1200,1] test <- zip.test[1:1000,-1] testC <- zip.test[1:1000,1]
いくつかのネットワークを並行してトレーニングする前に、正確に何を取得したいかを考える必要があります。 ニューラルネットワークの2つのパラメーター-隠れ層のニューロンの数とトレーニングの速度に興味があり、多かれ少なかれ最適な値を選択したいとします。 つまり、作業の結果は、ニューロンの数、学習速度、エラーの3つの数値を含むデータフレームになり、それに基づいてパラメーターの最適性が決まります。 前に、並列ループの結果をグループ化する関数について説明しました。 ベクトルよりも複雑な構造を持つデータの場合、そのような関数を自分で記述する必要があります。
comb <- function(df1, df2) if (df1$err < df2$err) df1 else df2
コードからわかるように、関数は2つのデータフレームのerrフィールドを比較し、エラーの少ないものを選択します。 パラレルモードでネットワークのトレーニングを直接提供するスクリプトフラグメントを以下に直接示します。 私たちはそれを起動し、しばらくの間、3つのカーネルの100%のロードを賞賛します。
errCl <- function (predicted, true){ return(round(sum(predicted != true)/length(true), 4)) } size <- c(13,29,53,71) lr <- c(.001,.01,.1,.5) nn.time <- system.time({ elm_par <- foreach(s=size, .combine='comb') %:% foreach(r=lr, .packages='RSNNS', .combine='comb', .inorder=F) %dopar% { elm <- elman(x=train, y=trainC, size=s, learnFunc="JE_BP", learnFuncParams=r, linOut=F, maxit=193) pred <- max.col(predict(elm, valid)) - 1 e <- errCl(pred, validC) data.frame(opt_size=s, lrate=r, err=e) } })
errCl関数は、分類エラーを決定するために使用されます。 サイズとlrベクトルには、それぞれ隠れネットワーク層のニューロン数とネットワーク学習率が含まれています。
%:%
命令を使用して、ネストされたループがどのように編成されているかに注目してください。 実際、詳細に入らない場合、通常、サイクルの本体でパラメーターを選択するときに起こることです。モデルはトレーニングデータセットでトレーニングされ、検証データセットが使用され、分類エラーが決定され、エラーの少ないパラメーターが選択されます。 私は次の結果を得ました:
> elm_par opt_size lrate err 1 29 0.5 0.05 > nn.time user system elapsed 0.312 0.168 179.542
もちろん、5%のエラーでお世辞を言わないでください。 テストセットで29個のニューロンと0.5のトレーニングレートでネットワークをテストしましょう。
elm <- elman(x=train, y=trainC, size=29, learnFunc="JE_BP", learnFuncParams=0.5, linOut=F, maxit=193) pred <- max.col(predict(elm, test)) - 1 e <- errCl(pred, testC)
> e [1] 0.119
まあ、11.9%も非常に良いです。 残っているのはクラスターを「返済する」ことだけです
stopCluster(cluster)
結論の代わりに
Rは非常に便利で陰湿な言語です。 それに、そして特にCRANのさまざまなパッケージに慣れているので、他のものに切り替えるのは難しいです。 しかし、もちろん、各言語には独自のニッチがあります。 Rの場合、これは統計およびデータ分析です。 また、Rは非常にゆったりしていますが、多くのタスクではそのパフォーマンスで十分です。
文学
Rによる高性能および並列コンピューティング
foreachループのネスト
RSNNSパッケージ