最大100ヌクレオチドの任意の実際に存在するプライマリシーケンスがあります。 形成する必要があるすべての水素結合を知っています。 出力は.pdbファイルです。このファイルでは、指定されたプライマリシーケンスの立体構造が使用され、必要なすべての水素結合が形成されます。
ここでは、誰もがそれが何であるかを試すことができるように、練習について話します。 私が話していることを計算するソフトウェアを開発しました。 ここで、 デモバージョンへのリンクを示します。 そして、私はあなたが見ることができるものを説明します。 100回聞くよりも1回見る方が良い:)
ダウンロード( ここからダウンロード )、解凍、RNAInSpace.exeの実行後、「開始」をクリックすると、画面が表示されます(空の上部ウィンドウと下部ウィンドウのみ)(既存のバグが既知で、上部ウィンドウが正常に配置されない場合があります-再起動して再試行してください):
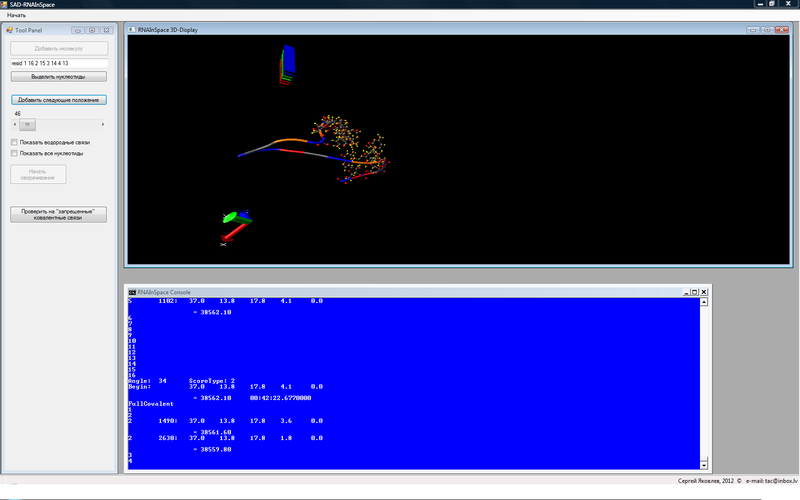
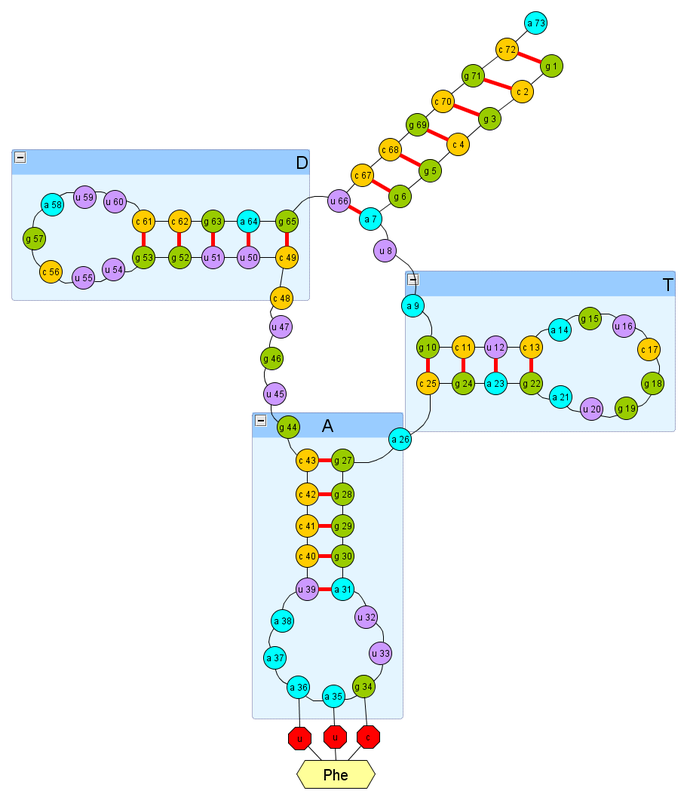
これは実際にはデモ版であり、折りたたみアルゴリズムを制御することはできません。 しかし、それ以外の場合は、折りたたみがどのように行われるかを追跡できます。 当初、このバージョンは、二次構造(「T」とラベルされた部分)の下にフェニルアラニンを保持する大腸菌の輸送RNAのTループの1つのヘリックスを折り畳むように構成されています。
次に、デモ版の管理方法。
ダウンロード後に最初に行うことは、「最小化の開始」ボタンをクリックすることです。 コンソールの下部画面に数字が表示されます。 それらについては後で。 RNAInSpace.exeが存在するディレクトリでファイルX _#。Pdbが表示され始めたので、X_0.pdbが表示されたらすぐに「分子の追加」ボタンをクリックします。 上部の画面では、RNAチェーンが一列に伸びています。 「ヌクレオチドの選択」ボタンをクリックできます。 相互に水素結合を形成するヌクレオチドのペアが4つあります(ただし、表示したい単語residの後に上記のヌクレオチド番号を入力することで変更できます)。 下の2つのチェックボックスは、水素結合(存在する場合)と鎖のすべてのヌクレオチドを表示/非表示します。
次のX_1.pdb、X_2.pdb、X_3.pdbなどが表示されるとすぐに。 [次の位置を追加]ボタンをクリックして、RNAがわずかに折り畳まれ始める様子を確認できます。
グラフィックウィンドウは展開できます。 マウスを制御するだけでなく、分子を回転させることができます(左/右ボタンを押して移動-回転、ホイールを回して増減します)。
プログラムをオフにして再起動すると、折り畳みは最初からではなく、前回折り畳みが停止した最後のステップから始まります。 最初から始めたい場合は、traj.datファイルをクリアする必要があります。 混乱しないように、次回起動するときには、古いX _#。PdbファイルとX _#。Txtファイルをすべて削除する必要があります。 PyMOLなどの標準的な分子イメージングソフトウェアで.pdb形式を表示することもできます。
デモ版はスパイラルが折り畳まれることを保証しません-これは幸運です:)
ビジュアルプレゼンテーションをいじって、計算中にコンソールの数字の意味を理解したい場合(過去の記事を注意深く読んでいると推測できますが)、デモプログラムで他に何ができるか、または質問するだけで、すぐに次のパートで試してみましょう質問をまとめてFAQを作成します。 もちろん、必要に応じて、関心があります。
PS誰かが幸運ではないのかもしれません。私のラップトップは、問題が何であるかを理解するまでグラフィックをロードしません。 あなたの問題について、可能な限り書きます。
更新しました。 このデモプログラムを同時にテストしています。 ステップ192(ファイルX_192.pdb)では、興味深い状況が表示されます。これについては、 パート5で説明しました。 1つの基本的な問題 。 らせんの内側にはまだ形成されていないが、らせんの端部(1-16ヌクレオチド)の間に水素結合が形成されています。 その後、それらは最初にループの近くで裂けて形成されます。