 現代のデータ分析の世界では、1つの方法または1つのアプローチのみを使用することは、遅かれ早かれ、あなたがどれだけ間違えたかという事実に出くわすことを意味します。 データ分析のためにさまざまな方法が組み合わされ、結果が比較され、比較に基づいてより正確な予測がすでに行われています。 ZINBAプログラムはまさにそのようなアプローチを使用しています。 開発者は、DNA seq実験を分析するためのさまざまな方法を1つのパッケージにまとめました。 このパッケージは、統計データ処理プログラムR用に作成されています。ZINBAは何をしますか? それらの一部が増幅された場合でも、たとえば化学的に、またはさまざまな程度の信号対雑音比を持っている場合でも、さまざまな濃縮領域を検出します。
現代のデータ分析の世界では、1つの方法または1つのアプローチのみを使用することは、遅かれ早かれ、あなたがどれだけ間違えたかという事実に出くわすことを意味します。 データ分析のためにさまざまな方法が組み合わされ、結果が比較され、比較に基づいてより正確な予測がすでに行われています。 ZINBAプログラムはまさにそのようなアプローチを使用しています。 開発者は、DNA seq実験を分析するためのさまざまな方法を1つのパッケージにまとめました。 このパッケージは、統計データ処理プログラムR用に作成されています。ZINBAは何をしますか? それらの一部が増幅された場合でも、たとえば化学的に、またはさまざまな程度の信号対雑音比を持っている場合でも、さまざまな濃縮領域を検出します。
最初は、 ZINBAソフトウェア製品に関するレビュー記事を作成する予定でしたが、使用されている方法について詳しく読むほど、アルゴリズムと定義を深く掘り下げます。 そして、将来、彼が得た知識を概説したとき、彼はデータがすでに複数の記事で収集されており、紹介なしでは問題の本質に触れることは難しいという事実に気づきました。 このトピックでは、ZINBAプログラムの説明で言及した記事から短い抜粋を提供し、自分のコメントで補足します。 私はあなたのコメントが真実の底に到達するのを一緒に待ちます。
規制生物学の広範な計画は、ゲノムが遺伝子発現のさまざまなメカニズムをどのようにエンコードするかを学ぶことです。 コーディングとこれらのメカニズムの関係は、クロマチン免疫沈降( ChIP )と遺伝子発現(RNA-seq)を使用して、ゲノム全体でタンパク質結合部位を見つける可能性によって明らかにされています。 証拠を探す必要はありません; DNA要素の百科事典プロジェクト(ENCODE [1])があり、その枠組みの中でゲノムの機能要素のほとんどが認識されています。 最初のステップはマイクロアレイ法を使用して行われましたが、ディープDNAシーケンシング(ChIP-seqおよびDNA-seq、時にはNGS次世代シーケンシングという用語が使用される)は、このテクノロジーに追いつき、追い越されました。
RNA-seqとChIP-seqには次のプラスの違いがあります。正確性、特異性、測定感度が向上し、ゲノム全体をすぐに調べることができます。 一般に、RNA-seqとChIP-seqは、マイクロチップなどの先行製品といくつかの類似点がありますが、詳細は大きく異なります。 しかし、シーケンシングには依然として多くの困難が伴いますが、これらの問題はマイクロチップよりもはるかに優れています。 切断されるDNAフラグメントがかなり短いという事実により、フラグメントはゲノム内で数回発生する可能性があり、DNAの反復部分の一部であるか、またはいくつかの開始部位の共通フラグメントであることが判明します。
データ処理では、ChIP反応を濃縮として考慮する必要があると考えられています。 通常、調製されたDNA溶液中の約60〜99%の断片がバックグラウンドであり、残りの1〜40%は、タンパク質が抗体を架橋できる断片に関連しているためです[2]。 シーケンス化されるのは、ソリューションのこの最小部分です。 濃縮度を計算するときは、汚染レベルが高いという事実を考慮する必要があります。 いくつかの場合、ゲノムあたりのリード数は比較にならないほど少ないです。たとえば、哺乳類ゲノムの場合、リードのリード数はゲノムの全長の1%未満です。 データのシーケンスには、明らかに新しいアルゴリズムとソフトウェアが必要です。
 図 1
図 1
このスライドは、ChIP-seqおよびRNA-seq分析の階層を模式的に示しています。 通常、分析はすべてのステップをボトムアップで行います。 回路の各レベルで異なるプログラムが適用され、ChIP-seqとRNA-seq専用のプログラムに時々分けられます。 あるプログラムの出力は、別のプログラムへの入力(パイプライン)です。 図からわかるように、すべてのプログラムは、転写産物のアセンブリを除き、ゲノムの読み取りの定義(マップ読み取り)を最初に通過します。 リードがゲノム上に配置された後、そのようなプログラムの出力は、Aggregate and identityプログラムの入力に送信されます。 たとえば、既知の注釈で濃縮領域を決定したり、密度を読み取ったりします。 また、これらのプログラムには、結合部位(DNA-seqの場合)と転写アイソフォームの新しい未探索フラグメント(ChIP-seqの場合)の両方の後続分析が含まれる場合があります。 以下は、より高いレベルの分析です。 それには、 保守的な動機や発現レベルの決定が含まれ、新しい遺伝子モデルの発見につながる可能性があります。 統合レベル(Integrate)は、現在のデータ処理では、以前に取得した結果を使用できることを示しています(必ずしも現在の実験、実験室、または国の一部としてではありません)。
それでは、ゲノム上でリードが見つかった後、私たちは何を得ますか? このデータはどのように見え、どのように互いに異なるのですか? DNAには1つまたは別のタンパク質が結合できる結合部位があり、これらの部位を結合部位または単に部位と呼びます。 たとえば、CTCFサイトとは、 CTCFタンパク質をこの領域に固定できることを意味します。 科学者は、CTCFタンパク質に対する抗体を発見し、その抗体を使用することで、対応するDNAのセクションを沈殿させることができました。 しかし、すでに知っているように、特別なセクションとともに、任意の抗体を抗体に付着させることができます。 他の方法も同様に機能します。タンパク質がDNAに結合し、抗体がタンパク質に結合し、DNAセクションの選択のみが変化します。 したがって、選択されたセクションの詳細はマークを残し、各メソッドの描画のランドスケープには独自の特徴があります。CTCFメソッドのように、どこか狭いピーク、グラフa。 例えば、RNAポリメラーゼII法、グラフbのように、隣接する領域が広い急峻なピーク。 ヒストン修飾が沈殿すると、連続的な「フリンジ」が得られます。これは、ヒストンの配置の特異性によるものです(図c)。 グラフでは、染色体上の座標がx軸に沿ってプロットされ、読み取りの密度がy軸に沿ってプロットされます。 図のデータは[4]から取得されます。
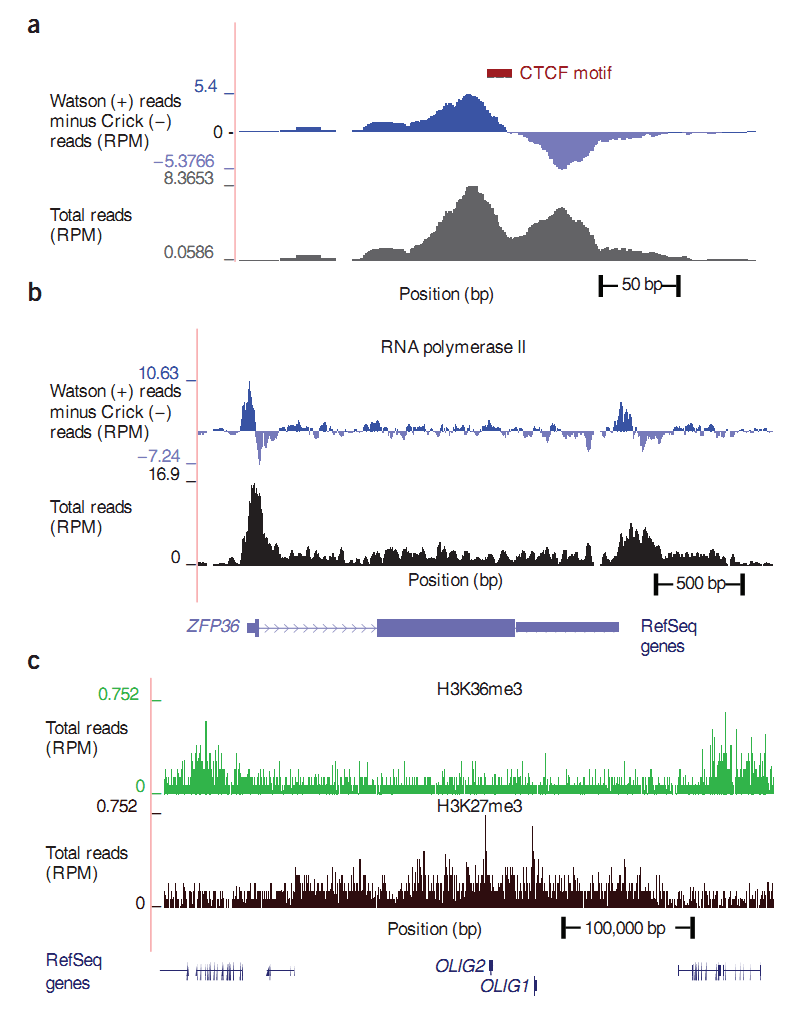 図 2
図 2
背景の存在により、アルゴリズムは「制御」に基づいて経験的にそれを評価しようとします。 制御は、特別な(免疫前の)抗体を使用した初期ソリューションで行われる追加の実験であり、何にも接続する必要はありませんが、ランダムに接続します。 この接続の結果は、任意の背景です。 一部のアルゴリズムは、受信したデータのみに基づいて、制御なしで、可能な背景をシミュレートします。 バックグラウンドレベルは、取得元の細胞と組織の種類、および堆積方法などに依存するため、どちらのアプローチを選択した場合でも(コントロールの有無にかかわらず)、バックグラウンドリードの分布を完全に統一することはできません。 。 増幅(PCR)およびシーケンシングに適用される技術は、人工濃縮(アーティファクト)を追加したり、あるセクションを別のセクションよりも増幅したりすることもできます。 各実験の充実した領域を見つけることができるアルゴリズムがあります。 各アルゴリズムは実験に対応するデータに対して先鋭化されていますが、アルゴリズムの基礎となる仮定は、DNA-seq [2]を使用して検出された濃縮領域の可能なセットに常に適合するとは限りません。
以下では、データを比較するために、ZIMBAに関する新しい記事のグラフがあります。 同様のグラフは、最初の図と2番目の図のスケールの違いによりぼやけていますが、DNAシーケンス技術に関する上記の説明と一致しているように見えます(図2を参照)。

図 3
前のグラフに加えて、図3は次のDNA-seq実験の状況を示しています:DNase感受性サンプル(DNase-seq)[5]およびホルムアルデヒドを使用したタンパク質の分離/固定(FAIRE-seq)[6]。
DNAは二重らせんです。 このフレーズ自体は人気がありますが、忘れられがちです。 私たちの場合、それはどういう意味ですか? そして、ゲノム上の読み取りを検索するプログラムが、染色体上の座標と、この読み取りがスパイラル(ストランド)のどちらの側であるかを示します。 スパイラルは、ポジティブ(センス)とノンポジティブ(ナンセンス/アンチセンス)に分けられます。 リードを取得するメカニズムは、ほとんどのリードがDNAに関連するタンパク質に対して2つの異なる側面に位置するようなものです。 さらに、ほとんどの場合、読み取りは対応するスパイラルの5 '端に配置されます。 したがって、両側の読み取りは、タンパク質結合部位の中心になる傾向があります。 図4はこれを模式的に示しています。赤い点は5 '端からの読み取りであり、中央はDNAに結合したタンパク質です。
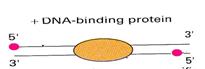
図 4
したがって、タンパク質-DNA結合部位の中心を見つけるために、次のグラフを作成します。 これを行うために、「y」軸で正と負の読み取り回数の差を先送りし、「x」軸で染色体に対応する読み取り座標を先送りします。 図5では、読み取りはプラス側に赤で、マイナス側に青で示されています。 結果として得られるグラフは、タンパク質結合部位の中心に非常に近いゼロと交差します(図5の下のグラフを参照)。 ヌクレオチドまで結合部位を決定するアルゴリズムが存在します。
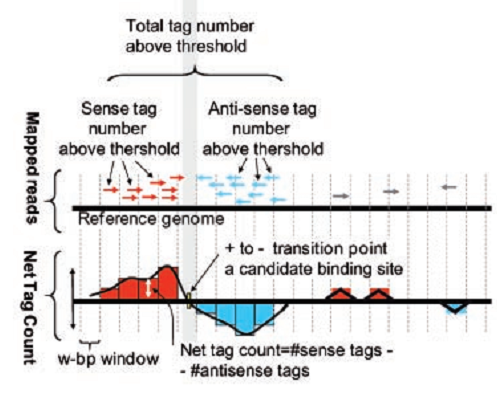
図 5
このトピックでは、研究の主題である資料を簡単に紹介しようとしました。 対応するDNA-seq実験のランドスケープの違いが示され、タンパク質結合部位の中心を近似的に見つける方法が説明されました。 巨大な仕事が待ち構えています:ZINBAの仕事についての議論を始めるためには、風景に依存するアルゴリズムの違いを理解し、ピークを見つけて、それらを人為的なものと区別し、ピークの近くにあるゲノムのどの領域が濃縮されていると考えられるかを見つけなければなりません。
1. Birney、E.、et al。、ENCODEパイロットプロジェクトによるヒトゲノムの1%の機能要素の同定と分析。 Nature、2007.447(7146):p。 799-816。
2. Pepke、S.、B。Wold、およびA. Mortazavi、ChIP-seqおよびRNA-seq研究用の計算。 Nat Methods、2009.6(11 Suppl):p。 S22-32。
3. Barski、A.およびK. Zhao、ChIP-Seqによるゲノム位置分析。 J Cell Biochem、2009.107(1):p。 11-8。
4. Barski、A.、et al。、ヒトゲノムにおけるヒストンメチル化の高解像度プロファイリング。 Cell、2007.129(4):p。 823-37。
5.ボイル、APおよびTSフレイ、クロマチンおよび遺伝子調節エレメントの高解像度マッピング研究。 エピゲノミクス、2009.1(2):p。 319-329。
6. Giresi、PGおよびJD Lieb、FAIRE(規制要素のホルムアルデヒド支援分離)を使用した、真核生物クロマチンからの活性調節要素の分離。 方法、2009.48(3):p。 233-9。
レビューは、オハイオ州シンシナティのアンドレイ・カルタショフによって作成されました。porter@ porter.st
Ekaterina Morozova、ekaterina @ porter.stにより修正。